4 Ergebnisse
4.1 Restriktionskartierung des pHD54 im Bereich von 'IS3Ea9'
4.2 Klonierungen für die vervollständigende Sequenzierung des 'IS3Ea9'
4.3 Sequenzierung von 'IS3Ea9'
4.4 Vergleichende Untersuchungen auf DNA- und Aminosäureebene
4.5 Vorkommen und Verbreitung von 'IS3Ea9'
Aufbauend auf Untersuchungen im 3'-Bereich des pEA9-nif-Clusters [Steibl, Dissertation (in Vorbereitung)] war es das Ziel der vorliegenden Diplomarbeit, das postulierte, neue IS-Element durch Sequenzierung genetisch zu charakterisieren und die mögliche Verbreitung in Enterobacter agglomerans 339, ggf. darüber hinaus, mittels DNA-DNA-Hybridisierungen zu untersuchen.
Für die Sequenzierung und Klonierung stand der Cosmidklon pHD54, der die vollständige nif-Region von pEA9 trägt [Steibl, 1989], und weitere Subklone, welche Restriktionsfragmente von pHD54 aus der 3'-flankierenden Region des nif-Clusters enthalten, zur Verfügung: Die Klonierung des 5.4 kb HindIII-BamHI-Fragmentes in die HindIII- und BamHI-Schnittstelle von pUC18 ergab den Klon pJJ5.4HB und durch Klonierung des 0.8 kb SalI-BamHI-Fragmentes in die entsprechenden Schnittstellen von pUC18 wurde pJJ0.8SB hergestellt [Steibl, Dissertation (in Vorbereitung)].
Die vollständige Sequenzierung des IS-Elementes wurde mit doppelsträngiger Plasmid-DNA nach der Methode von Sanger durchgeführt [Sanger et al., 1977]. Als Sequenziervektor wurde pUC18 verwendet, was die Sequenzierung beider DNA-Stränge eines Inserts durch Verwendung von zwei verschiedenen Primern ermöglichte.
4.1 Restriktionskartierung des pHD54 im Bereich von 'IS3Ea9'
Aus Sequenzdaten war bereits bekannt, daß das 0.8 kb SalI-BamHI-Fragment den Großteil des IS-Elementes trägt; auch einige Restriktionsschnittstellen waren schon in diesem Bereich kartiert [Steibl, Dissertation (in Vorbereitung)]. Doch um für die spätere Klonierung von Restriktionsfragmenten zur anschließenden Sequenzierung eventuell geeignetere Schnittstellen zu finden, wurde die bereits bestehende Restriktionskarte vom rechten Randbereich des pEA9-nif-Clusters durch eine Nachkartierung mit weiteren Restriktionsenzymen ergänzt und über den bisher kartierten Bereich hinaus erweitert. Im Vordergrund stand dabei die Suche nach Fragmenten, die möglichst große Teile des IS-Elementes tragen. Da es sich bei pHD54 um ein Plasmid von annähernd 47 kb handelte, dessen Restriktionsverdau eine unüberschaubare Vielzahl von Fragmenten ergeben hätte, mußten die in diesem Zusammenhang interessanten Restriktionsfragmente markiert werden. Diese Markierung zur vereinfachten Restriktionsanalyse erfolgte mittels Hybridisierung, was die Herstellung einer geeigneten DNA-Sonde erforderlich machte. Um eine gezielt 'IS3Ea9'-spezifische DNA-Sonde herzustellen, die sich auch für spätere Untersuchungen zur Bestimmung von Kopienzahl und Verbreitung wiederverwenden läßt, wurde Plasmid-DNA von pJJ0.8SB, der das 0.8 kb SalI-BamHI trägt, im Restriktionsverdau mit BamHI und SalI geschnitten und im Agarose-Gel elektrophoretisch aufgetrennt. Nach Elution des 0.8 kb SalI-BamHI-Fragmentes und entsprechender Aufreinigung erfolgte die Markierung mit Digoxigenin-11-dUTP, wie unter 3.10.2.1 beschrieben. Die Kontrolle der Konzentration markierter DNA erfolgte mit Dot Blot, wie unter 3.10.2.2 erläutert, und zeigte eine gute Ausbeute von 1 ng Dig-markierter DNA/µl 'Labeling'-Ansatz. Das entsprechend markierte 0.8 kb SalI-BamHI-Fragment aus pJJ0.8SB wurde im folgenden stets als 0.8 SB-Sonde bezeichnet.
Zur Kartierung wurde nun Plasmid-DNA von pHD54 mit verschiedenen Restriktions-enzymen in Einzel-und Doppelverdaus geschnitten. Der Verdau mit HpaI und SphI sollte dabei neue, noch nicht kartierte Schnittstellen aus dem 3'-Bereich von pEA9 zeigen, während das Restriktionsmuster mit PstI- und SalI-Verdaus zum einen den Anschluß an die bestehende Restriktionskarte von pHD54, zum anderen aber auch weitere Schnittstellen im noch nicht kartierten Bereich liefern sollte. Nach Auftrennung der pHD54-Restriktions-verdaus mittels Agarose-Gelelektrophorse und Übertragung auf eine Nylonmembran mittels Southern Blot, erfolgte die Hybridisierung des Filters mit der 0.8 SB-Sonde unter hoher Stringenz wie unter 3.10 beschrieben.
Das Ergebnis ist in Abbildung 5 dargestellt. Im Detail zeigten dabei folgende Restriktionsfragmente ein Hybridisierungssignal (in der Reihenfolge: Spur 1-9, Abb.5): 1.9 kb PstI/HpaI, 3.9 kb PstI, 0.9 kb PstI/SphI, 3.8 kb SphI, 3.5 kb SphI/HpaI, 4.5 kb HpaI, 2.0 kb HpaI/SalI, 6.5 kb SalI und 1.0 kb SalI/SphI.
Die Auswertung dieses Restriktions- bzw. Hybridisierungsmusters ermöglichte zunächst die Kartierung von drei neuen Restriktionsschnittstellen im 3'-Bereich des pEA9-nif-Clusters: Zum einen befinden sich im 1.4 kb BamHI-Fragment von pHD54 eine HpaI- und SphI-Schnittstelle in asymmetrischer Anordnung, wobei die SphI-Schnittstelle im Abstand von ca. 0.8 kb in 5'-Richtung von der HpaI-Schnittstelle liegt. Desweiteren konnte auf dem 5.9 kb BamHI-Fragment eine weitere SphI-Schnittstelle zwischen der kartierten HindIII- und HpaI-Schnittstelle im Abstand von 0.4 kb zur HpaI-Schnittstelle nachgewiesen werden. Durch die Größe der hybridisierenden SalI- und PstI-Fragmente konnte außerdem die Orientierung des 42.4 kb-Inserts im pHD54 bestimmt werden, da die im 3'-Bereich gefundene PstI- und SalI-Schnittstelle bereits außerhalb des pEA9-Inserts im pJA1-Vektor kartierte. Die erweiterte Plasmidkarte von pHD54 ist in Abbildung 6 dargestellt.
Abbildung 5: Restriktionsverdau von pHD54 mit Hybridisierungsergebnis
_________________________________________________________________________

Spur 1: pHD54 + PstI + HpaI Spur 6: pHD54 + HpaI
Spur 2: pHD54 + PstI Spur 7: pHD54 + HpaI + SalI
Spur 3: pHD54 + PstI + SphI Spur 8: pHD54 + SalI
Spur 4: pHD54 + SphI Spur 9: pHD54 + SalI + SphI
Spur 5: pHD54 + SphI + HpaI Spur 10: KBL
_________________________________________________________________________
Abbildung 5: Links das Bandenmuster der pHD54-Restriktionsverdaus mit KBL-Längenstandard, rechts der entsprechende Filter nach Hybridisierung mit 0.8 SB-Sonde. Die Hybridisierungssignale sind mit den jeweiligen Fragmentgrößen beschriftet, die Gel-Banden mit Hybridisierungssignal durch Pfeile gekennzeichnet. Nummern unter dem Gel bzw. Filter geben die jeweilige Spur an.
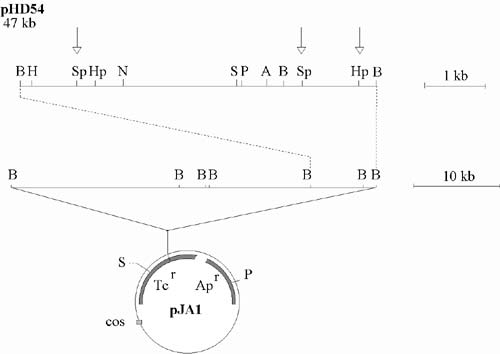
Abbildung 6: Detailausschnitt der Plasmidkarte von pHD54
_________________________________________________________________________
_________________________________________________________________________
Abbildung 6: Plasmidkarte von pHD54 mit pJA1 als ringförmiger Vektor und das BamHI-Insert im Maßstab 10 kb als Winkel dargestellt. Die 5.9 und 1.4 kb-BamHI-Fragmente des Inserts sind als Ausschnitt im Maßstab 1 kb vergrößert (oben). Die neu kartierten Restriktionsschnittstellen sind mit einem Pfeil markiert. Restriktionsenzyme sind wie folgt abgekürzt: A-ApaI, B-BamHI, H-HindIII, Hp-HpaI, N-NotI, P-PstI, S-SalI und Sp-SphI. Die kartierten Schnittstellen für BglII und KpnI wurden der Übersichtlichkeit halber nicht dargestellt.
Der pHD54-SphI-Restriktionsverdau zeigte im Agarose-Gel bei 3.8 kb eine potentielle Doppelbande (Abb.5, Spur 4, mit Pfeil markiert), was zunächst nur anhand der Leuchtintensität dieser Bande im UV-Licht auffiel: Es bestand die Möglichkeit, daß an dieser Stelle bei 3.8 kb zwei Fragmente gleicher Größe im Gel laufen. Da dieses SphI-Fragment für eine spätere Klonierung interessant war, wurde die Möglichkeit einer Doppelbande genauer untersucht. Den Nachweis sollte ein Vergleich mit pHD21 erbringen: Die klonierten pEA9-Inserts von pHD54 und pHD21 überlappen in einem Bereich von 35.1kb, jedoch fehlt pHD21 der Randbereich von pHD54 mit dem 5.9 und 1.4 kb-BamHI-Fragment und damit auch das 3.8 kb SphI-Fragment A im IS-homologen Bereich. Existierten tatsächlich zwei 3.8 kb SphI-Fragmente, so wird Fragment B bei pHD54 durch A 'überdeckt', während B bei pHD21 sichtbar sein mußte. Nach Restriktionsverdau von pHD54 und pHD21 mit SphI und elektrophoretischer Auftrennung zeigte pHD21 im Gegensatz zu pHD54 keine 3.8 kb SphI-Doppelbande (Ergebnis nicht abgebildet), womit die Existenz von zwei SphI-Fragmenten gleicher Größe auf pHD54 nachgewiesen war.
4.2 Klonierungen für die vervollständigende Sequenzierung des 'IS3Ea9'
Klonierungsstrategie
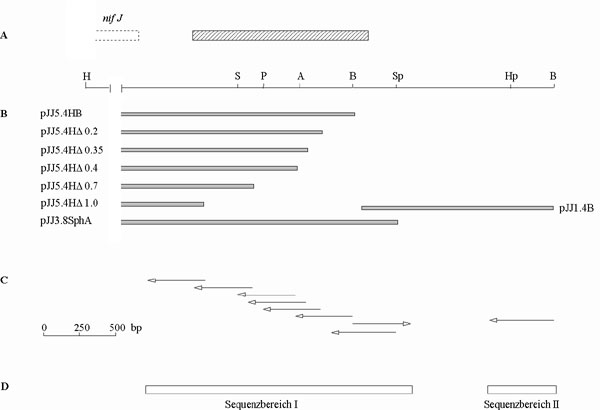
Mit den Ergebnissen der Ansequenzierung von pJJ0.8SB stand fest, daß Teile des IS-Elementes, bei einer vergleichbaren Größe der IS3-Elemente von etwa 1.2 kb, sowohl in 5'-Richtung von der SalI-Schnittstelle, als auch in 3'-Richtung von der BamHI-Schnittstelle liegen mußten [Steibl, Dissertation (in Vorbereitung)]. Damit erstreckte sich das postulierte IS-Element über den im pJJ5.4HB subklonierten Bereich auf das 1.4 kb BamHI-Fragment hinaus. Die Herstellung von Deletionsklonen des pJJ5.4HB, die Klonierung des 1.4 kb BamHI-Fragmentes aus pHD54 mit anschließender Sequenzierung würde somit die vollständige Sequenz im Einzelstrang liefern.
Ausgehend von der erweiterten Restriktionskarte des pHD54 bot sich für die Sequenzierung des Gegenstranges die Klonierung des 3.8 kb SphI-Fragmentes an. Eine Übersicht der Klonierungsstrategie ist in Abbildung 7 wiedergegeben.
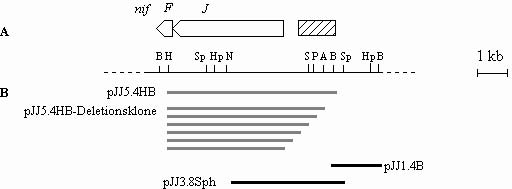
Abbildung 7: Übersicht der Klonierungsstrategie
_________________________________________________________________________
_________________________________________________________________________
Abbildung 7: A) Ausschnitt aus der Restriktionskarte von pEA9 in 5'-3'-Richtung im Maßstab 1 kb. Die großen offenen Pfeile kennzeichnen die Lage und Orientierung der Gene nifF und nifJ im 3'-Bereich des nif-Clusters. Die vermutete Lage des IS-Elementes ist mit einer schraffierten Box markiert. Restriktionsenzyme sind wie folgt abgekürzt: A-ApaI, B-BamHI, H-HindIII, Hp-HpaI, N-NotI, P-PstI, S-SalI und Sp-SphI. Die kartierten Schnittstellen für BglII und KpnI wurden der Übersichtlichkeit halber nicht dargestellt.
B) Die schwarzen Balken kennzeichnen zu klonierende, die grauen Balken klonierte Bereiche in der IS-homologen Region; die Beschriftung gibt die Bezeichnung der jeweiligen Klone an.
4.2.1 Herstellung der Deletionsklone von pJJ5.4HB
Um den IS-homologen Bereich auf pJJ5.4HB vollständig zu sequenzieren, wurde dieses Plasmid einer ExonukleaseIII-Behandlung unterzogen.
PJJ5.4HB Plasmid-DNA wurde mit BamHI und SstI als Schutzgruppe geschnitten; anschließend erfolgte die ExonukleaseIII-Behandlung wie unter 3.4.3 beschrieben. Dabei wurde das 5.4 kb HindIII-BamHI-Insert von der BamHI-Seite abgebaut, was die erwünschten Insertdeletionen erzeugte (Abb. 8 u. 9). Nach Transformation der ligierten Reaktionsansätze verschiedener Zeitstufen in E.coliDH5a-Zellen wurden die jeweiligen Transformationsansätze auf LB-Ap-Medien ausplattiert. Von den etwa 20 Transformanten je Platte wurden pro Zeitwert fünf Klone ausgewählt. Plasmid-DNA dieser ausgewählten Transformanten wurde mit HindIII linearisiert und gelelektrophoretisch auf die Deletionsgröße überprüft. Von den insgesamt 50 Klonen wurden letztlich fünf, welche Deletionen im Bereich von 0.2 bis 1.0 kb zeigten, ausgewählt und für die spätere Sequenzierung verwendet.
Die Klone wurden entsprechend ihrer Deletionsgröße als pJJ5.4HD0.2, pJJ5.4HD0.35, pJJ5.4HD0.4, pJJ5.4HD0.7 und pJJ5.4HD1.0 bezeichnet (Abb 8 u. 9).
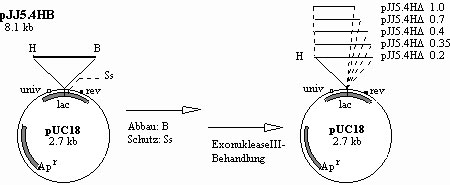
Abbildung 8: ExonukleaseIII-Abbau von pJJ5.4HB
_________________________________________________________________________
_________________________________________________________________________
Abbildung 8: Der ExonukleaseIII-Abbau ist anhand der Plasmidkarten von pJJ5.4HB und den hergestellten Deletionsklonen schematisch dargestellt. Der jeweilige Insertbereich der Plasmide ist als Balken vom pUC18-Vektor abgehoben. Zur besseren Übersicht wurden lediglich die für die ExonukleaseIII-Behandlung relevanten Restriktionsschnittstellen abgebildet. Die Restriktions-enzyme wurden wie folgt abgekürzt: B-BamHI, H-HindIII und Ss-SstI.
Abbildung 9: Restriktionsverdau von pJJ5.4HB und Deletionsklonen
_________________________________________________________________________
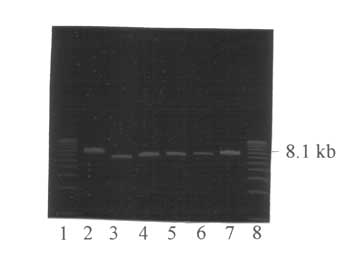
Spur 2: pJJ5.4HB + HindIII Spur 6: pJJ5.4HD0.35 + HindIII
Spur 3: pJJ5.4HD1.0 + HindIII Spur 7: pJJ5.4HD0.2 + HindIII
Spur 4: pJJ5.4HD0.7 + HindIII Spur 8: KBL
_________________________________________________________________________
Abbildung 9: Bandenmuster von pJJ5.4HB und ausgewählten Deletionsklonen nach Linearisierung mit HindIII. Die Größe von pJJ5.4HB ist mit 8.1 kb gekennzeichnet.
4.2.2 Herstellung von pJJ1.4B
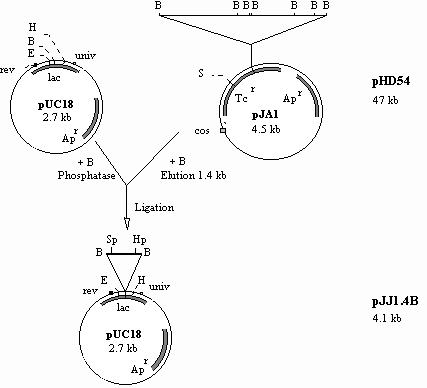
Zunächst wurde pUC18-DNA mit BamHI linearisiert und stand nach Dephoshorylierung als Vektor für die anschließende Klonierung zur Verfügung. Zur Präparation des späteren Inserts wurde pHD54-DNA mit BamHI geschnitten und gelelektrophoretisch aufgetrennt. Nach Elution und Aufreinigung des 1.4 kb BamHI-Fragmentes erfolgte die Ligation mit dem vorbereiteten pUC18-Vektor (Schema der Klonierung in Abb.10).
Abbildung 10: Klonierung von pJJ1.4B
_________________________________________________________________________
_________________________________________________________________________
Abbildung 10: Klonierung des 1.4 kb BamHI-Fragments von pHD54 in die BamHI-Schnittstelle von pUC18. Oben sind die Plasmide pUC18 (Vektor) und pHD54 (mit 1.4 kb BamHI-Fragment) dargestellt, unten das Klonierungsprodukt pJJ1.4B nach Restriktionskartierung. Zur besseren Übersicht wurden nur die für die Klonierung und Orientierung notwendigen Restriktionsschnittstellen abgebildet. Die entsprechenden Restriktionsenzyme wurden wie folgt abgekürzt: B-BamHI, E-EcoRI, H-HindIII, Hp-HpaI, S-SalI und Sp-SphI.
Der Ligationsansatz wurde anschließend in E.coliDH5a-Zellen transformiert und zur a-Komplementation auf LB-Ap-Platten mit IPTG-/XGal-Zusatz ausplattiert. Von den 20 lacZ--Transformanten wurden zehn ausgewählt, die durch Restriktionsverdau mit BamHI auf das Vorhandensein des erwarteten 1.4 kb BamHI-Inserts gescreent wurden. Von den acht Klonen, die ein entsprechendes Insert zeigten, wurde einer für die spätere Sequenzierung ausgewählt und als pJJ1.4B bezeichnet. Zunächst wurde der ausgewählte Klon jedoch einer detaillierten Restriktionskartierung unterzogen (Abb.11):
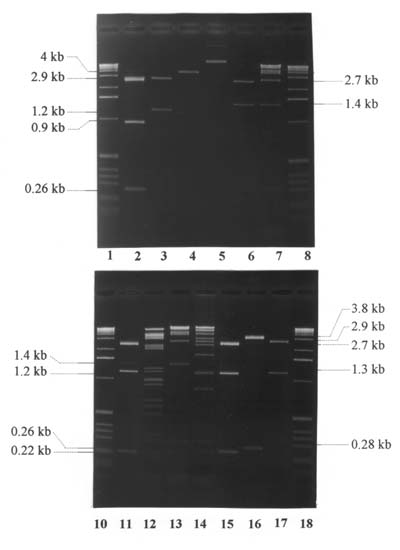
Durch Restriktionsverdau von pHD54 und pJJ1.4B mit BamHI ließ sich in beiden Fällen das identische 1.4 kb Fragment zeigen. Als zweite Bande zeigte pJJ1.4B die erwartete 2.7 kb Vektor-Bande des pUC18. Durch Restriktionsverdau mit HpaI und SphI sollte die Anzahl und der Abstand entsprechender Schnittstellen im pJJ1.4B bestimmt werden, da gerade diese Schnittstellen unter 4.1 im pHD54 kartiert wurden. DNA von pJJ1.4B wurde ungeschnitten und im Restriktionsverdau mit beiden Enzymen im Einzel- und Doppelverdau im Gel aufgetrennt. Der HpaI-Verdau ergab ein 4 kb Fragment und damit eine Linearisierung des Plasmides, zeigte also nur eine Schnittstelle. Da jedoch im pUC18 keine HpaI-Schnittstelle vorkommt, mußte diese im Insert liegen. Der Verdau mit SphI ergab Fragmente von 2.9 und 1.2 kb und damit auch zwei Schnittstellen im pJJ1.4B, von denen eine in der 'Polycloning-Site' des pUC18 liegt. Im Doppelverdau mit HpaI und SphI zeigten sich Banden bei 2.9, 0.9 und 0.26 kb, d.h., das 1.2 kb SphI-Fragment wurde durch HpaI in ein 0.9 und 0.26 kb Fragment geschnitten.
Zur Bestätigung der Orientierung des Inserts im pJJ1.4B wurde der Abstand der HpaI-Schnittstelle zur externen EcoRI-und HindIII-Schnittstelle im Vektor bestimmt. Der Doppelverdau mit HpaI und EcoRI zeigte ein 2.9 und 1.3 kb Fragment. Restriktionsverdau mit HpaI und HindIII ergab dagegen ein 3.8 und 0.28 kb Fragment. Damit stand die Orientierung fest: Die HpaI- liegt dicht zur HindIII-Schnittstelle im pUC18, während die SphI-Schnittstelle im Abstand von ca. 0.9 kb zur HpaI- in Richtung der EcoRI-Schnittstelle des Vektors liegt. Um nachzuweisen, daß das 1.4 kb BamHI-Fragment identisch zu dem im pJJ1.4B ist, wurden beide Plasmide in Doppelverdaus mit BamHI/HpaI, BamHI/SphI und einmal pHD54 nur mit BamHI (zur besseren Übersicht im Gel) geschnitten. Im BamHI/HpaI-Verdau zeigten sich für beide Plasmide die jeweils gleichen Banden bei 1.2 und 0.26 kb; die BamHI/SphI-Verdaus beider Plasmide ergaben die identischen Banden bei 0.22 und 1.2 kb. Das Ergebnis belegte eindeutig, daß das 1.4 kb Insert im pJJ1.4B dem 1.4 kb BamHI-Fragment von pHD54 entspricht.
Das vollständige Ergebnis der Restriktionskartierung wurde in Abbildung 11, die abgeleitete Plasmidkarte von pJJ1.4B bereits in Abbildung 10 dargestellt.
Abbildung 11: Restriktionskartierung von pJJ1.4B
_________________________________________________________________________
Spur 2: pJJ1.4B + HpaI + SphI Spur 11: pJJ1.4B + BamHI + SphI
Spur 3: pJJ1.4B + SphI Spur 12: pHD54 + BamHI + SphI
Spur 4: pJJ1.4B + HpaI Spur 13: pHD54 + BamHI
Spur 5: pJJ1.4B (ungeschnitten) Spur 14: pHD54 + BamHI + HpaI
Spur 6: pJJ1.4B + BamHI Spur 15: pJJ1.4B + BamHI + HpaI
Spur 7: pHD54 + BamHI Spur 16: pJJ1.4B + HpaI + HindIII
Spur 8: KBL Spur 17: pJJ1.4B + HpaI + EcoRI
Spur 18: KBL
_________________________________________________________________________
Abbildung 11: Restriktionskartierung von pJJ1.4B und pHD54 zum Nachweis der Klonierung des 1.4 kb BamHI-Fragmentes und Bestimmung der Orientierung des Inserts im pJJ1.4B durch Kartierung der asymmetrischen HpaI- und SphI-Restriktionsschnittstellen. Die Größe wichtiger Fragmente ist markiert, Erläuterungen befinden sich im Text.
4.2.3 Herstellung von pJJ3.8SphA
Zur Sequenzierung des Gegenstranges im Übergangsbereich der Subklone pJJ5.4HB und pJJ1.4B wurde das 3.8 kb SphI-Fragment, welches bei der Kartierung des pHD54 ein Hybridisierungssignal gab, in die SphI-Schnittstelle von pUC18 kloniert.
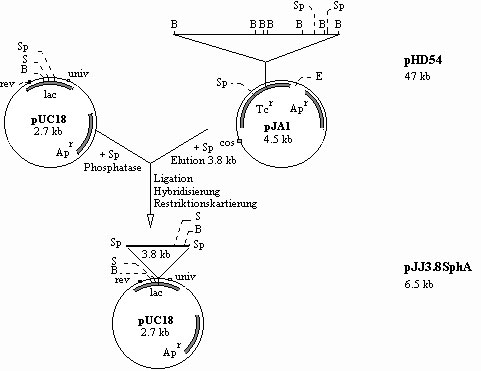
Zur Klonierung wurde pUC18-DNA durch Restriktionsverdau mit SphI und anschließende Dephosphorylierung als Vektor vorbereitet. Für die Präparation des 3.8 kb SphI-Inserts zeigte sich allerdings das Problem zweier gleich großer SphI-Fragmente im pHD54. Für die Klonierung bedeutete dies praktisch, zunächst das 3.8 kb 'SphI-Fragmentgemisch' zu klonieren und später auf den gesuchten Klon mit dem gewünschten SphI-Fragment zu screenen.
Abbildung 12: Klonierung des pJJ3.8SphA
_________________________________________________________________________
_________________________________________________________________________
Abbildung 12: Herstellung des pJJ3.8SphA duch Klonierung des 5'- 3.8 kb SphI-Fragmentes von pHD54 in die SphI-Schnittstelle von pUC18. Die Plasmide pUC18 (Vektor) und pHD54 (mit 3.8 kb SphI-Fragmenten) sind oben abgebildet, unten das Klonierungsprodukt pJJ3.8SphA. Zur besseren Übersicht wurden nur die für die Klonierung und Kartierung notwendigen Restriktionsschnittstellen eingezeichnet; die Restriktionsenzyme wurden wie folgt abgekürzt: B-BamHI, E-EcoRI, S-SalI und Sp-SphI.
Ein Restriktionsverdau von pHD54 mit SphI wurde im Agarose-Gel aufgetrennt, die 3.8 kb SphI-Doppelbande eluiert, das 'Fragmentgemisch' mit dem vorbereiteten pUC18 Vektor ligiert und in E.coliDH5a-Zellen transformiert. Der Transformationsansatz wurde auf LB-Ap-Platten mit IPTG-/X-Gal-Zusatz ausplattiert. Von zehn weißen lacZ--Kolonien wurde Plasmid-DNA gewonnen, mittels Dot Blot auf eine Nylonmembran übertragen und mit der 0.8 SB-Sonde hybridisiert. Von den vier Klonen, die ein Hybridisierungssignal gaben, wurde einer ausgewählt und als pJJ3.8SphA bezeichnet. Von den sechs Klonen, die kein Hybridisierungssignal zeigten, wurden zwei aufbewahrt, jedoch nicht weiter untersucht. Sie wurden als pJJ3.8SphB bezeichnet. Im Anschluß erfolgte eine Restriktionskartierung von pJJ3.8SphA (Abb. 13).
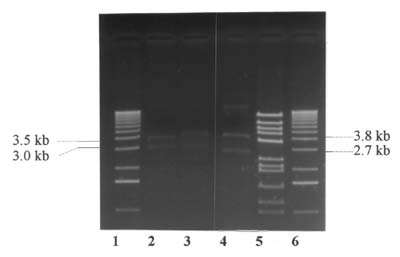
Das Restriktionsmuster zeigt im SphI-Verdau von pJJ3.8SphA und pHD54 das klonierte 3.8 kb Fragment. Im SalI-Verdau von pJJ3.8SphA konnte durch ein 3.8 kb Fragment die Orientierung des Inserts im Vektor bestimmt werden, denn diese 3.8 kb müssen sich aus den Einzelgrößen von pUC18 (2.7 kb) und dem kartierten SalI-SphI-Fragment (1 kb; beschrieben unter 4.1) zusammensetzen. Die Fragmentengrößen im BamHI-Verdau von pJJ3.8SphA zeigten Abstände von 3.0 kb bzw. 3.5 kb für die zwei BamHI-Schnittstellen. Da eine davon an bekannter Stelle im Vektor liegt, mußte die zweite im Insert im Abstand von 3.5 kb zur ersten liegen. Damit stand die Orientierung des Inserts fest und das entsprechende Kartierungsergebnis ist in Abbildung 12 dargestellt.
Der hergestellte Klon pJJ3.8SphA ist für weitere Untersuchungen an 'IS3Ea9' interessant, da nun erstmals das vollständige IS-Element in einem Vektor subkloniert vorliegt.
Abbildung 13: Restriktionskartierung von pJJ3.8SphA
_________________________________________________________________________
 Spur 1: KBL Spur 4: pJJ3.8SphA + SphI
Spur 1: KBL Spur 4: pJJ3.8SphA + SphI
Spur 2: pJJ3.8SphA + BamHI Spur 5: pHD54 + SphI
Spur 3: pJJ3.8SphA + SalI Spur 6: KBL
_________________________________________________________________________
Abbildung 13: Bandenmuster zur Kartierung von pJJ3.8SphA nach Restriktionsverdau. Die relevanten Restriktionfragmente wurden entsprechend ihrer Größe gekennzeichnet. Erläuterungen im Text.
4.3 Sequenzierung von 'IS3Ea9'
4.3.1 Sequenzierungsstrategie
Zur Bestimmung der DNA-Sequenz des IS-Elementes im Einzelstrang sollten pJJ5.4HB, pJJ5.4HB-Deletionsklone und pJJ1.4B sequenziert werden. Zur Sequenzierung des Gegenstranges sollte pJJ3.8SphA, bzw. dessen Deletionsklone, verwendet werden.
Da die Sequenzierung mit dem M13-reverse Primer von pUC18 im Einzelstrang bereits große Überlappungen für den Bereich der pJJ5.4HB-Deletionsklone und ein soweit eindeutiges Sequenzergebnis erbrachte, wurde auf die Herstellung von pJJ3.8SphA-Deletionsklonen verzichtet. Durch Ansequenzierung des pJJ3.8SphA unter Verwendung des M13-universal Primers wurde der Gegenstrang im Bereich des pJJ1.4B bestimmt. Die schematisierte Sequenzierungsstrategie ist in Abbildung 14 dargestellt.
Abbildung 14: Sequenzierungsstrategie
_________________________________________________________________________
_________________________________________________________________________
Abbildung 14: A) Detailausschnitt der Restriktionskarte von pEA9. Das Gen nifJ ist als offene, punktierte Box und die Position des IS-Elementes als schraffierte Box abgebildet. Restriktionsenzyme sind wie folgt abgekürzt: A-ApaI, B-BamHI, H-HindIII, Hp-HpaI, P-PstI, S-SalI und Sp-SphI. Die kartierten Schnittstellen für BglII und KpnI wurden nicht dargestellt. B) Zur Sequenzierung verwendete Klone wurden als graue Boxen abgebildet. C) Die Pfeile symbolisieren die Ausdehnung der Sequenzierreaktionen der einzelnen Klone. D) Die offenen Boxen kennzeichnen die durch Sequenzierung erhaltenen zusammenhängenden Sequenzbereiche (I u.II).
Die Sequenzierung des pJJ1.4B erfolgte sowohl mit 'universal' als auch mit 'reverse' Primer um durch den Nachweis der SphI- und HpaI-Schnittstelle in den jeweiligen Randbereichen die Orientierung des 1.4 kb Inserts parallel zur Restriktionskartierung zu belegen. Dem sequenzierten Randbereich mit der HpaI-Schnittstelle entspricht der in Abbildung 15 dargestellte Sequenzbereich II.
4.3.2 Sequenzanalyse von Sequenzbereich II
4.3.2.1 Nukleotid- und abgeleitete Aminosäuresequenz
Im Sequenzbereich II wurde über einen Bereich von 479 bp die Nukleotidsequenz im Einzelstrang bestimmt. Die ermittelte Basenfolge mit abgeleiteten AS-Sequenzen ist in Abbildung 15 gezeigt (Positionsangaben beziehen sich immer auf die dargestellte Nukleotidsequenz).
Abbildung 15: Nukleotid- und abgeleitete AS-Sequenz von Sequenzbereich II
_________________________________________________________________________
ORFaðïORFb
1 CATATAAGCGACGAGCAACCTTCCTAACTGGTACGTCTGATGAACAGGCGAAATAGATGT 60
ORFa V Y S L L V G R I D H A D Y L S A L S T
ORFb K D A K D V
ORFað
61 TTCATCTATCAGGCCGATATCGCGACGAGTATAAAAAGTTAGTCGTGGAATTAGAAAAAG 120
ORFa K
ORFb F Y I T R S Y R Q E Y K E I L V K I K K
121 GTGACTTTATGGTTATGGTCATTGCGGCTATTTCAGTGGAAATATCAGTAACTGGAATAC 180
ORFb W Q F V L V L L A S L T V K I T M S R I
HpaI ïORFb ŽSD
181 CAATTGCTTATGTAGTTCTACCCATACTAGGAGACCTAGACCTGCCTGTAGTTAGCTTAT 240
ORFb T L S Y M
241 CCCAAACAGGTGCCCGTCGCGAAATTGGTCCTTCTTGGTTGCCAGGGCATCTGAGAGAAG 300
ïORFc
301 TCTCTGACAAAGCTCCTATTGGTCCTTCTTGGTTGCCAGGGCATCTGAGAGAAGTCTCTG 360
ORFc L
361 ACAAGCTCCACGCAAACCCCAGTATCATTGCCATCCAAAATAGTCTTTGCTTTATCCTTT 420
ORFc S N S T R K P D Y Y R Y T K D S V F Y S
BamHI
421 TTTATCATAGCTTTGCTACCAGATAAAGCAACTCCATTGCTTAGTAACGTAGTCCTAGGA 479
ORFc F I T D F R H D I E N L Y R I M A D P D
ïORFc
_________________________________________________________________________
Abbildung 15: Ermittelte DNA-Sequenz über einen 479-bp-Bereich von pEA9; angegeben ist nur die Sequenz in 5'-3'-Richtung. Die aus der Nukleotidsequenz abgeleiteten AS-Sequenzen für ORFa, ORFb und ORFc sind unterhalb der Nukleotidsequenz mit Einbuchstabensymbolen angegeben; es wurden nur ORFs mit einer Mindestlänge von 40 aa berücksichtigt. Shine-Dalgarno-Sequenzen (SD) sind mit einer Linie gekennzeichnet, Start- und Stoppcodons jeweils fett gedruckt. Erkennungsstellen für Restriktionsenzyme sind durch horizontale Linien über der DNA-Sequenz markiert.
4.3.2.2 Auswertung von Sequenzbereich II
Bei der Untersuchung der Nukleotidsequenz von Sequenzbereich II, für die ein GC-Gehalt von 44 % berechnet wurde, zeigte sich in 3'-Richtung ein offenes Lesesraster, bezeichnet als orfa. Die Untersuchung des Gegenstranges in 5'-Richtung ergab die offenen Leseraster orfb und orfc.
Orfa läuft vom 5'-Ende außerhalb der Sequenz bis zu Position 64. Im sequenzierten Bereich umfaßt dieses Leseraster demnach 21 aa. Von Nukleotid 194 bis 42 liegt orfb, das für 51 aa kodiert. Orfc erstreckt sich vom 3'-Ende außerhalb des sequenzierten Bereiches bis zu Nukleotid 356 und codiert in diesem Bereich für 21 aa. Datenbankrecherchen anhand der aufgeführten offenen Leseraster ergaben jedoch keine Homologien mit bereits sequenzierten Genen. Darum wurde an dieser Stelle die Auswertung von Sequenzbereich II abgeschlossen.
4.3.3 Sequenzanalyse von Sequenzbereich I
4.3.3.1 Nukleotid- und abgeleitete Aminosäuresequenz
Im Sequenzbereich I wurde über einen Bereich von 1969 bp die Nukleotidsequenz im Einzelstrang bestimmt (im Doppelstrang von Position 1550 bis 1969). Die ermittelte Basenfolge mit abgeleiteten AS-Sequenzen ist in Abbildung 16 gezeigt. Spätere Positionsangaben beziehen sich immer auf die in dieser Abbildung dargestellte Nukleotid-sequenz.
Abbildung 16: Nukleotid- und abgeleitete AS-Sequenz von Sequenzbereich I
_________________________________________________________________________
1 GTGTCTATCAGGAGGGTTTGTCGGAATTGAATGTTTCGCCACACATTTCTTAGCAACTTG 60
61 TCGCTACACTGACATTATCCGCTCCGATTTAGGAACTTATCCACTCAGGTTTTCAGACTG 120
121 TAGTAAAGCTGATGTCTTTTTATGCCTGAAATTCAGCCCGGCAAAATATGGCCAGTACGG 180
181 TTTTTCTCAACGGGGTTTTAGAAATCCAGCTCAAGTGCAAAACACAGGTAGGACATCAGC 240
241 GGCGCGCAATGCCTGAACTGCTGTGCGCAAAAATCCATAACATCCGGCTTGCACAGATCC 300
301 TCAAACGCAATGCTTTTAACCGAGATAAAATCTTTGCGTTTTGATTCCGCTAACAACAGA 360
IHF
IR-L -35
361 ATCTGTTGCTAACATTACTGAAGATCCTCAGAGTGACCTGCTCCCAGTTGATTAGTACAC 420
ACTGGACGAGGGTCAACTAATCATGTG
-10 0 SD ORFAð
421 CCCGATGTTAGTAATGTCTTCATAAGCCACATGAGGACATCCCCATGAAGAAGCGTTTTT 480
GGGCTACAATCATTACAGAAGTATTCGGTGTACTCCTGTAGGGGTACTTCTTCGCAAAAA
ORFA M K K R F S
Chi
481 CCGACCAACAGATCATCAGTATTCTCCGCGAGGCTGAAGCCGGGGTATCCGCCCGTGAAC 540
GGCTGGTTGTCTAGTAGTCATAAGAGGCGCTCCGACTTCGGCCCCATAGGCGGGCACTTG
ORFA D Q Q I I S I L R E A E A G V S A R E L
541 TCTGCCGCAAGCACGCCATTTCCGATGCCACGTTTTACACCTGGCGTAAGAAGTATGGCG 600
AGACGGCGTTCGTGCGGTAAAGGCTACGGTGCAAAATGTGGACCGCATTCTTCATACCGC
ORFA C R K H A I S D A T F Y T W R K K Y G G
601 GTATGGAGGTGCCTGAGGTTAAGCGCCTGAAGTCGCTTGAGGAAGAGAACGCCAGACTCA 660
CATACCTCCACGGACTCCAATTCGCGGACTTCAGCGAACTCCTTCTCTTGCGGTCTGAGT
ORFA M E V P E V K R L K S L E E E N A R L K
DnaA
661 AGAAGCTGCTTGCCGAAGCCATGCTGGATAAAGAGGCGCTTCAGGTGGCTCTTGGGCGAA 720
TCTTCGACGAACGGCTTCGGTACGACCTATTTCTCCGCGAAGTCCACCGAGAACCCGCTT
ORFA K L L A E A M L D K E A L Q V A L G R K
ORFAð SD ORFBð
721 AGTACTGACGACAGACCAGAAGCGGGAAGCCGTGATGTTGATATGTGATGCGACCGGTCT 780
TCATGACTGCTGTCTGGTCTTCGCCCTTCGGCACTACAACTATACACTACGCTGGCCAGA
ORFA Y
ORFB M L I C D A T G L
PstI SalI
781 GTCGCAACGTCGTGCCTGCAGGCTCACAGGTTTATCTCTGTCGACCTGCCGCTATGAGGC 840
CAGCGTTGCAGCACGGACGTCCGAGTGTCCAAATAGAGACAGCTGGACGGCGATACTCCG
ORFB S Q R R A C R L T G L S L S T C R Y E A
PstI
841 TCAGCGCCCGGCTGCAGATGCGCATTTATCAGGGCGCATCACTGAGCTGGCACTGGAGCG 900
AGTCGCGGGCCGACGTCTACGCGTAAATAGTCCCGCGTAGTGACTCGACCGTGACCTCGC
ORFB Q R P A A D A H L S G R I T E L A L E R
901 CAGGCGTTTTGGCTACCGTCGCATCTGGCAGTTGCTGCGCCGTGAAGGGCTACATGTTAA 960
GTCCGCAAAACCGATGGCAGCGTAGACCGTCAACGACGCGGCACTTCCCGATGTACAATT
ORFB R R F G Y R R I W Q L L R R E G L H V N
ïORFC
961 TCATAAGCGCGTGTACAGGCTTTATCACCTCAGTGGCCTGGGCGTAAAACGCAGAAGACG 1020
AGTATTCGCGCACATGTCCGAAATAGTGGAGTCACCGGACCCGCATTTTGCGTCTTCTGC
ORFB H K R V Y R L Y H L S G L G V K R R R R
ORFC R L P R P T F R L L R
1021 TCGTAAAGGGCTGGCAACAGAACGTCTCCCGCTGCTCCGTCCGGCGGCACCCAATCTGAC 1080
AGCATTTCCCGACCGTTGTCTTGCAGAGGGCGACGAGGCAGGCCGCCGTGGGTTAGACTG
ORFB R K G L A T E R L P L L R P A A P N L T
ORFC R L P S A V S R R G S S R G A A G L R V
1081 CTGGTCTGATGATTTCGTCATGGACGCATTGGCCACCGGTCGCAGGATCAAGTGCCTTAC 1140
GACCAGACTACTAAAGCAGTACCTGCGTAACCGGTGGCCAGCGTCCTAGTTCACGGAATG
ORFB W S D D F V M D A L A T G R R I K C L T
ORFC Q D S S K T M S A N A V P R L I L H R V
1141 CTGCGTAGACGACTTCACGAAGGAATGCCTGACGGTCACTGTTGCCTTTGGGATTTCAGG 1200
GACGCATCTGCTGAAGTGCTTCCTTACGGACTGCCAGTGACAACGGAAACCCTAAAGTCC
ORFB C V D D F T K E C L T V T V A F G I S G
ORFC Q T S S K V F S H R V T V T A K P I E P
ïORFC SD
1201 CGTGCAGGTCACGCGTATTCTGGACAGCATTGCGCTGTTTCGCGGCTATCCGGCGACGAT 1260
GCACGTCCAGTGCGCATAAGACCTGTCGTAACGCGACAAAGCGCCGATAGGCCGCTGCTA
ORFB V Q V T R I L D S I A L F R G Y P A T I
ORFC T C T V R I R S L M
0 ApaI -10 -35
1261 AAGAACGGATCAGGGCCCGGAATTTACCTGCCGCGCACTCGATCAATGGGCCTTTGAGCA 1320
TTCTTGCCTAGTCCCGGGCCTTAAATGGACGGCGCGTGAGCTAGTTACCCGGAAACTCGT
ORFB R T D Q G P E F T C R A L D Q W A F E H
DnaA
1321 TGGCGTGGAGCTGCGACTTATCCAGCCCGGCAAGCCGACACAGAACGGATTTATTGAGAG 1380
ACCGCACCTCGACGCTGAATAGGTCGGGCCGTTCGGCTGTGTCTTGCCTAAATAACTCTC
ORFB G V E L R L I Q P G K P T Q N G F I E S
1381 TTTTAACGGACGCTTTCGCGATGAATGCCTGAATGAACACTGGTTCAGTGACGTCAGTCA 1440
AAAATTGCCTGCGAAAGCGCTACTTACGGACTTACTTGTGACCAAGTCACTGCAGTCAGT
ORFB F N G R F R D E C L N E H W F S D V S H
1441 TGCCAGGAAAACCATCAGTGAATGGCGTCAGGATTATAATGAGTACCGCCCGCACTCCGC 1500
ACGGTCCTTTTGGTAGTCACTTACCGCAGTCCTAATATTACTCATGGCGGGCGTGAGGCG
ORFB A R K T I S E W R Q D Y N E Y R P H S A
1501 GCTGAATTATCAGGCGCCGTCTGAATTTGCGGCGGCCTGGAGAAAGGGTAATTCTGATAG 1560
CGACTTAATAGTCCGCGGCAGACTTAAACGCCGCCGGACCTCTTTCCCATTAAGACTATC
ORFB L N Y Q A P S E F A A A W R K G N S D S
BamHI ORFBð IR-R
1561 TGAAGGATCCGACATTACTAAGTGAGCGTTGTATCTAATCCTGGGGGCAGGTCAATTGGC 1620
ACTTCCTAGGCTGTAATGATTCACTCGCAACATAGATTAGGACCCCCGTCCAGT
ORFB E G S D I T K
ïORFD
1621 GTGTGCCGGGCAGCAATTAACATTAGCTGATAATATGCAGTATTTGAGGCAAATCGCCGC 1680
ORFD S I I H L I Q P L D G
1681 TGATGACACCTAGAATTACAGTGTTTTCACATAGTGAGGTAAATCGCCTTCTTATGTAGA 1740
ORFD S I V G L I V T N E C L S T F R R R I Y
SphI ïORFD
1741 AACGATCAGTATCAGCATCGCGGCAGCCCATCACTCCGCTGCATGCCGGATTATTCATCC 1800
ORFD F R D T D A D R C G M V G S C A P N N M
ïORFE
1801 ATTTTTACGACACAATTCCGCCCCTGCCGCTTGGCTTGATATAAAGCAATATCTGCGCGC 1860
ORFE S V I G G R G S P K I Y L L I Q A
ïORFFïORFG
1861 TCCACCAAGGACTCAGTTCCTCATTGCCTTGGGTCTCTGCAATTCCACAGCTTATGGTGA 1920
ORFE S W W P S L E E N G Q T E A I G C S I T
ORFF Q R P D R C N W L K H H
ORFG P S
1921 TCGGTATGGTTTGCTTCTGAGATACGTGATGTGCATGTGCCGACAGGTC 1969
ORFE I P I T Q K Q S V H H A H A S L D
ORFF D T H N A E S I R S T C T G V P
ORFG R Y P K S R L Y T I H M H R C T
_________________________________________________________________________
Abbildung 16: Ermittelte Nukleotidsequenz von Sequenzbereich I über einen 1969 bp-Bereich von pEA9 in 5'-3'-Richtung. Die DNA-Sequenz im Bereich von 'IS3Ea9' ist doppelsträngig dargestellt, die Randbereiche im Einzelstrang. Terminale 'Inverted Repeats' (IR-L u. IR-R), Start- und Stoppcodons sind fett gedruckt. Sequenzen mit Homologie zur -10- und -35-Region der Consensussequenz von E.coli -Promotoren sind mit einer horizontalen Linie und -10/-35 gekennzeichnet. Shine-Dalgarno-Sequenzen (SD) und potentielle Bindungsstellen für IHF und DnaA sind mit einer horizontalen Linie markiert, ebenso wie ein Bereich mit Homologie zu Chi-Sequenzen. Die abgeleiteten AS-Sequenzen sind unterhalb der Nukleotidsequenz mit Einbuchstabensymbolen angegeben; es wurden nur vollständige ORFs mit über 40 aa und ansequenzierte ORFs mit über 10 aa berücksichtigt. Erkennungsstellen für Restriktionsendonukleasen sind durch horizontale Linien über der DNA-Sequenz markiert.
Die Regionen von Position 1-703, 820-1125 und 1301-1575 entsprechen den von Steibl sequenzierten und analysierten Bereichen. Ein Vergleich dieser Abschnitte mit der von Steibl ermittelten Sequenz ergab keine Abweichungen. Die Analysen im Bereich von Position 1 bis 394 sind Bestandteil der Dissertation von Steibl (in Vorbereitung). Aus diesem Grunde wurden alle folgenden Untersuchungen basierend auf der vollständigen Sequenz ausschließlich in dem für das IS-Element relevanten Bereich bzw. darüber hinaus nur in 3'-Richtung von Position 394 bis 1969 durchgeführt.
4.3.3.2 Offene Leseraster
Die Sequenz wurde in beide Richtungen auf das Vorhandensein von offenen Leserastern untersucht. Hierbei konnten für Sequenzbereich I insgesamt sieben offene Leseraster, davon vier vollständig, gefunden werden (Tabelle 6).
Tabelle 6: Offene Leseraster im Sequenzbereich I
_________________________________________________________________________
|
|
Bezeichnung des offenen Leserasters |
Position [bp] |
Größe [aa] |
_________________________________________________________________________
|
orfA * |
465 - 725 |
87 |
||
|
orfB * |
755 - 1582 |
276 |
||
|
orfC * |
1230 - 988 |
81 |
||
|
orfD * |
1798 - 1646 |
51 |
||
|
orfE # |
(1969) - 1808 |
(54) |
||
|
orfF # |
(1969) - 1884 |
(29) |
||
|
orfG # |
(1969) - 1915 |
(18) |
_________________________________________________________________________
Tabelle 6: Vollständige offene Leseraster wurden mit * markiert. Bei offenen Leserastern mit der Kennzeichnung # wurden lediglich terminale Bereiche ansequenziert; die Größe wurde deshalb in Klammern angegeben.
Datenbankrecherchen mit den vier offenen Leserastern im 3'-Randbereich, orfD, orfE, orfF und orfG, ergaben jedoch keine Homologien zu bereits sequenzierten Genen. Aus diesem Grunde wurde die weitere Untersuchung dieser Region eingestellt.
Im Bereich von 394 bis 1614, dem IS-Element, wurden die drei offenen Leseraster orfA, orfB und orfC gefunden, wobei orfC innerhalb von orfB auf dem Gegenstrang liegt. Zur Prüfung, ob es sich bei diesen offenen Leserastern um kodierende DNA-Bereiche handelt, wurde der gesamte Sequenzbereich I in beide Richtungen mit Hilfe des GCG-Programms TESTCODE untersucht. Das Programm nutzt die Tatsache, daß an der dritten Triplettposition bei kodierenden DNA-Sequenzen bestimmte Nukleotide mit gleichbleibender Häufigkeit eingebaut werden [Fickett, 1982]. Die Auswertung für den Vorwärtsstrang zeigte, daß zwischen den Positionen 500-700 bzw. 900-1650 (jeweils ± 200bp) mit hoher Wahrscheinlichkeit kodierende Sequenzen vorliegen. Die Lage dieser kodierenden Bereiche entsprach der Position von orfA und orfB. Die Untersuchung des Gegenstranges zeigte ebenfalls eine hohe Wahrscheinlichkeit für eine kodierende Sequenz im Bereich von orfC.
Desweiteren wurde die Sequenz vor den offenen Leserastern auf das Vorhandensein einer ribosomalen Bindungsstelle hin untersucht, da diese eine Voraussetzung für die Initiation der Translation ist. Ribosomale Bindestellen sind gekennzeichnet durch ein Startcodon (AUG) und die SD-Sequenz [Shine u. Dalgarno, 1975], die wenige Basen 'stromaufwärts' vor dem Startcodon liegt. Die Effizienz einer ribosomalen Bindungsstelle ist abhängig von der Länge der SD-Sequenz, dem Abstand dieser Sequenz vom Startcodon und der relativen Lage innerhalb einer möglichen Sekundärstruktur der mRNA [Gold, 1988].
Es konnten sowohl vor orfA, als auch vor orfB solche ribosomalen Bindungsstellen gefunden werden; orfC zeigte nur ein sehr schwaches Motiv. Ein Vergleich mit der SD-Consensus-Sequenz wurde in Tabelle 7 aufgeführt.
Tabelle 7: Vergleich potentieller SD-Sequenzen
_________________________________________________________________________
SD-Sequenz Abstand zum AUG [bp]
SD-Consensus-Sequenz 5'- AGGAGGU -3' 4 - 9
orfA 5'- AGGACAU -3' 4
orfB 5'- GGGAAGC -3' 4
orfC 5'- CGGAUAG - 3' 16
_________________________________________________________________________
Tabelle 7: Vergleich der Translationsstartstellen von orfA, orfB und orfC mit der SD-Consensus-Sequenz. Die fett gedruckten Basen stimmen mit der Consensus-Sequenz überein.
Nach weiteren Analysen zeigten sich zwei potentielle Promotor-Strukturen vor orfA und orfC, die Homologien zu s70-Promotoren von E.coli zeigten: P1 von Position 406 bis 443 und P2 von 1269 bis 1308. Ein Vergleich der -35, -10 und 0-Region dieser Bereiche mit der Promotor-Consensus-Sequenz ist in Tabelle 8 aufgeführt.
Tabelle 8: Vergleich von Promtorstrukturen
_________________________________________________________________________
Promotor -35-Region -10-Region 0-Region
s70 TCTTGAC TATAAT CAT
P1 AGTTGAT TAGTAAT CAT
P2 CATTGATC TAAATT GAT
_________________________________________________________________________
Tabelle 8: Beis70 handelt es sich um die Consensus-Sequenz vons70-Promotoren von E.coli. P1 bezeichnet den vor orfA, P2 den vor orfC gefundenen Promotor (P1 postuliert von Steibl, s. Einleitung). Übereinstimmende Nukleotide zur Consensus-Sequenz wurden fett gedruckt. Mögliche Einschübe von Nukleotiden wurden durch eine Unterstreichung markiert. Die Angaben für die Consensus-Sequenz erfolgten nach Lewin [Lewin, 1987].
Neben der starken Sequenzhomologie zeigten die Promotorstrukturen P1 und P2 jedoch auch die -35/-10 -Abstände zum Transkriptionsstart bei Position 0, wie sie für die Consensus-Sequenz beschrieben sind [Lewin, 1987]. Die Wahrscheinlichkeit, daß es sich bei P1 und P2 um tatsächliche Promotoren für einen Transkriptionstart handelte, war demnach sehr hoch.
4.3.3.3 IS-Strukturen
Die Region von Nukleotid 394 bis 1614 im Sequenzbereich I unterscheidet sich in ihrem GC-Gehalt von den flankierenden Bereichen:
Während für die Randbereiche von Position 1-394 und 1614-1969 GC-Gehalte von 43 % (links) bzw. 48 % (rechts) berechnet wurden, ergab sich für die mittlere Region ein deutlich höherer GC-Gehalt von 56 %. Desweiteren wird der Bereich von Position 394-1614 durch unvollständige 'Inverted Repeats' von 46 bp Länge eingerahmt, die insgesamt zehn Fehlpaarungen aufweisen. Bei der vorliegenden Struktur handelte es sich um das vollständige, von Steibl [Dissertation (in Vorbereitung)] postulierte IS-Element der IS3-Familie.
Für viele IS-Elemente und auch innerhalb der IS3-Familie, wurden eine Reihe von Sequenzmerkmalen beschrieben, deren genaue Funktion noch nicht bekannt ist, von denen aber angenommen wird, daß sie u.a. für die Regulation der Transposition von Bedeutung sind. Entsprechend den jeweiligen Literaturangaben wurde die Sequenz von 'IS3Ea9' nach charakteristischen Merkmalen durchsucht:
a) Neben den bereits lokalisierten 'Inverted Repeats' wurde direkt angrenzend an diese Bereiche nach flankierenden 'Direct Repeats' (DR), auch bezeichnet als 'Target Duplikationen', gesucht. Es konnten jedoch keine direkten Sequenzwiederholungen gefunden werden.
b) Für eine Reihe von IS-Elementen ließen sich Bindestellen für DnaA nachweisen, ein für die Initiation der Chromosomenreplikation essentielles Protein [Kornberg, 1980]. Für IS50 konnte in vitro die Erkennung und Bindung von DnaA an spezifische Sequenzen gezeigt werden [Fuller et al., 1984] und entsprechend ließ sich in vivo eine Beeinflussung der Aktivität von IS50 nachweisen [Phadnis u. Berg, 1985; Yin u. Reznikoff, 1987]. Das Vorhandensein von DnaA Bindestellen ist inzwischen für viele IS-Elemente, wie z.B. IS1, IS150 und IS3411, belegt; über die funktionelle Bedeutung herrscht jedoch Unklarheit. In 'IS3Ea9' fanden sich an Position 682 und 1338 zwei Bereiche starker Homologie zu der Consensus-Sequenz AATAGGTGT für DnaA-Bindestellen bei E.coli [Galas u. Chandler, 1989], bei denen es sich höchstwahrscheinlich auch um entsprechende Bindungsstellen handelt.
c) Ein weiteres häufig gefundenes Sequenzmotiv sind IHF-Bindestellen. Für IS1 ist bekannt, daß IHF in vitro an die Erkennungssequenz im IR-L bindet und durch sterische Veränderungen der Sekundärstruktur die Termini von IS1 gegen DNaseI-Abbau schützt [Prentki, 1987b; Gamas et al., 1987]. Bei Tn1000 verstärkt IHF die Bindung der Transposase an die DNA; die Folge ist eine erhöhte Transpositionsrate [Grindley u. Wiater, 1988]. In 'IS3Ea9' befindet sich eine mögliche IHF-Bindestelle bei Position 408, direkt im -35-Bereich des Promoters im IR-L und zeigt eine hohe Homologie zu der Consensus-Sequenz AAnnnn TTGAT [Galas u. Chandler, 1989].
d) Bei Chi-Sequenzen handelt es sich um DNA-Bereiche, welche die RecBCD-Endonuklease aktivieren; somit sind sie als 'hot-spots' für homologe Rekombination von großer Bedeutung [Smith, 1988]. Das Vorkommen von Chi-Sequenzen wurde bereits in IS186 nachgewiesen [Galas u. Chandler, 1989]. Die Suche nach möglichen Chi-Sequenzen in der Nukleotidsequenz von 'IS3Ea9' zeigte an Position 482 ein wahrscheinliches Motiv mit hoher Homologie zur Chi-Consensus-Sequenz CGACCACC.
e) Eine Untersuchung von 'IS3Ea9' auf Dam-Methylierungssequenzen zeigte keine Entsprechung zum GATC-Consensus. Erkennungssequenzen für die Dam-Methylase finden sich allerdings in einer großen Zahl von IS-Elementen [Galas u. Chandler, 1989], denn gerade die Methylierung von Promotorbereichen ermöglicht eine Regulation der Transposition in Kopplung an die Replikation des Wirts-Replikons: Transient hemimethylierte DNA-Stränge bewirken eine verstärkte Transposase-Expression und Transpositionsrate. Nachgewiesen wurde dieser Mechanismus für IS50 [Krebs u. Reznikoff, 1986] und IS10 [Roberts et al., 1985].
Einen autoregulatorischen Effekt haben Sekundärstrukturen auf Ebene der mRNA, wobei die Translation von transkribierten IS-Genen, beispielsweise orfA, verhindert wird. Sekundärstrukturen der mRNA machen bei IS10 die Shine-Dalgarno-Sequenz (SD), das Signal der Translationsinitiation, unzugänglich und verhindern somit die Translation [Trieu-Cuot u. Courvalin, 1984]. Auch für IS150 wurde ein entsprechender Schutzmechanismus über Sekundärstrukturen beschrieben [Schwartz et al., 1988].
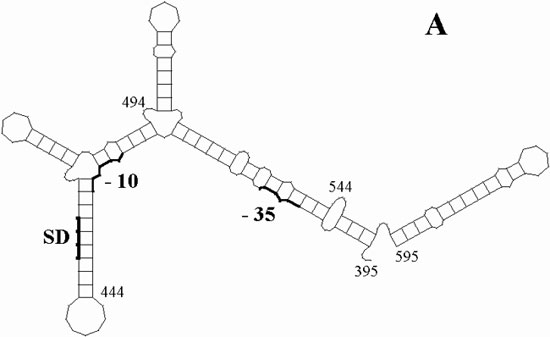
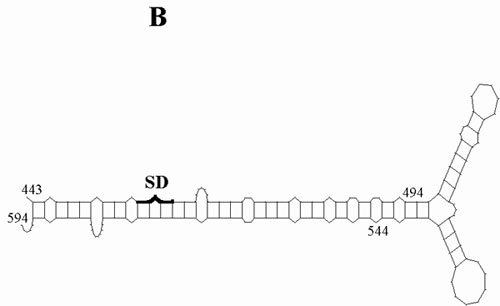
Bei der Untersuchung von 'IS3Ea9' auf das Vorhandensein derartiger Mechanismen wurden um den Transkriptionsstart bei Promotor P1 (Position 434-460) wie bei IS150 [Schwartz et al., 1988] 'Inverted Repeats' gefunden. Um die möglichen Auswirkungen auf die Sekundärstrukturen einer mRNA abzuschätzen, wurden zunächst zwei Möglichkeiten durchgespielt: Fall A - von einem externen Promotor wird in das IS-Element hinein transkribiert, d.h., beide 'Repeats' liegen auf der mRNA; Fall B - die Transkription beginnt innerhalb des IS-Elementes am internen Promoter bei Position 443 und nur das 3'-'Repeat' wird transkribiert. Unter Verwendung des GCG-Programms FOLD/SQUIGGLES [Zuker, 1981] wurde für beide Möglichkeiten jeweils eine mögliche Sekundärstruktur entwickelt. Die entsprechende Graphik ist in Abbildung 17 dargestellt. Um einen Anhaltspunkt für konkrete Aussagen in Bezug auf diese Sekundärstrukturen zu finden, wurde das Beispiel von IS150 herangezogen, da für dieses IS-Element ein Einfluß von Sekundärstrukturen in der Literatur beschrieben ist. Für IS150 wurden ebenfalls die beiden Möglichkeiten der Transkription durchgespielt: Es wurde für die Transkription von einem externen und vom internen Promotor vor orfA die jeweilige mRNA ebenfalls mit Hilfe des Programms FOLD gefaltet, wobei die gleichen Parameter wie bei den Sekundärstrukturen in Abbildung 17 verwendet wurden. Die resultierenden Sekundärstrukturen (Ergebnis nicht dargestellt) zeigten ein zu 'IS3Ea9' vergleichbares Bild.
Abbildung 17: Potentielle mRNA-Sekundärstrukturen für 'IS3Ea9'
_________________________________________________________________________
_________________________________________________________________________
Abbildung 17: Mögliche Sekundärstrukturen der mRNA sind für einen Bereich von 200 bp dargestellt; SD kennzeichnet die Shine-Dalgarno-Sequenz von orfA, als -35 u. -10 wurden Bereiche von Promotor P1 markiert. A: Transkriptionsstart liegt außerhalb des IS-Elementes; B: Transkriptionsstart am internen Promotor (bei Position 443); Erläuterung im Text. Die Abbildung wurde mit Hilfe des GCG-Programms FOLD/SQUIGGLES [Zuker, 1981] erstellt.
Aus der Abbildung wird deutlich, daß die beiden Annahmen A und B zwei unterschiedliche Strukturen produzieren. Ein konkreter Einfluß, beispielsweise der Lage der SD-Sequenz innerhalb eines stabilen 'Stemloops', läßt sich zwar nicht ableiten, doch deuten die Strukturen A und B allein durch ihre Unterschiedlichkeit auf einen Mechanismus, unter Umständen, wie er für IS10 und IS150 beschrieben wurde: Um den Transkriptionsstart am IS-internen Promoter liegen Inverted Repeats, von denen bei autoregulierter Transkription nur das vom Promoter aus 'downstream' gelegene Repeat abgelesen wird. Erst bei von außen einlaufender Transkription werden beide Repeats abgelesen und können Sekundärstrukturen in Form von Haarnadelschleifen bilden, die die SD-Sequenz von orfA unzugänglich machen könnten.
4.3.3.4 Zusammenfassung der Sequenzanalyse
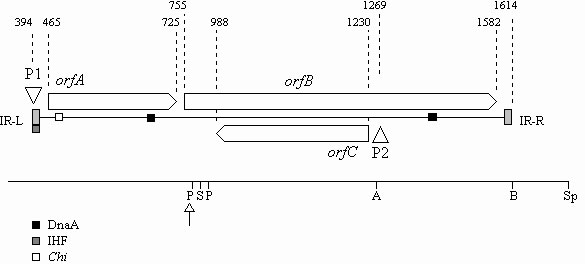
In einer schematisierten Übersicht wurden die einzelnen Bestandteile des sequenzierten IS-Elementes zusammengefaßt; die Übersicht ist in Abbildung 18 graphisch dargestellt.
Abbildung 18: Strukturübersicht für die DNA-Sequenz von 'IS3Ea9'
_________________________________________________________________________
_________________________________________________________________________
Abbildung 18: In dieser schematischen Übersicht wurden die Strukturmerkmale des IS-Elementes 'IS3Ea9' zusammengefaßt. Oben sind die offenen Leseraster als offene Pfeile, IR als graue Boxen und die Promotoren P1 u. P2 als Dreiecke dargestellt. Mögliche Bindestellen für DnaA, IHF sowie Chi-Sequenzen sind wie angegeben gekennzeichnet. Die jeweiligen Positionsangaben wurden oberhalb der Grafik angegeben, unterhalb befindet sich die entsprechende Restriktionskarte für diesen Bereich. Restriktionsenzyme wurden wie folgt abgekürzt: A-ApaI, B-BamHI, P-PstI, S-SalI u. Sp-SphI. Die mit einem Pfeil markierte PstI-Schnittstelle zeigte sich erst durch die Sequenzierung; aufgrund des geringen Abstandes von 50 bp zur kartierten PstI-Schnittstelle konnte ein Nachweis durch Restriktionsverdau noch nicht erbracht werden.
4.4 Vergleichende Untersuchungen auf DNA- und Aminosäureebene mit Merkmalen anderer IS-Elemente der IS3-Familie
Das bei E.aggl.339 gefundene IS-Element zeigte in Datenbankrecherchen stets hohe Homologien zu IS-Elementen der IS3-Familie. Im folgenden sollen nun einige Merkmale des neu gefundenen IS-Elementes mit Eigenschaften bereits beschriebener IS-Elemente verglichen werden. Zu diesem Zweck wurden aus dem Verwandtschaftsbereich der IS3-Familie einige Vertreter ausgewählt, die stellvertretend für weitere Untergruppen hoher Sequenzhomologie stehen. Für alle folgenden Alignments und Vergleiche wurden die in Tabelle 9 aufgeführten IS-Elemente herangezogen, die jedoch allesamt der IS3-Familie angehören.
Tabelle 9: Insertionssequenzen der IS3-Familie
_________________________________________________________________________
IS Herkunft Referenz Länge [bp]
_________________________________________________________________________
|
IS3 |
Escherichia coli |
Timmermann u. Tu, 1985 |
1 258 |
|
IS150 |
Escherichia coli |
Schwartz et al.,1988 |
1 443 |
|
1 IS3411 |
Escherichia coli |
Ishiguro u. Sato, 1988 |
1 310 |
|
IS861 |
Streptococcus B COH-1 |
Rubens et al.,1989 |
1 442 |
|
1 ISR1 |
Rhizobium lupini |
Priefer et al., 1989 |
1 259 |
|
IS911 |
Shigella dysenteriae |
Prère et al., 1990 |
1 250 |
|
1 IS476 |
Xanthomonas campestris |
Kearney u. Staskawicz, 1990 |
1 226 |
|
IS407 |
Pseudomonas cepacia |
Wood et al., 1991 |
1 236 |
|
'IS3Ea9' |
Enterobacter agglomerans 339 |
diese Arbeit |
1 221 |
_________________________________________________________________________
Tabelle 9: Die veröffentlichten Sequenzen der mit 1 markierten IS-Elemente wurden entsprechend den Angaben nach Prère geändert [Prère et al., 1990]: IS476 wurde eine Base hinzugefügt (ein C nach Nukleotid 221), ebenfalls IS3411 (ein T nach Nukleotid 938). Bei ISR1 wurden zwei Basen entfernt (Nukleotid 461 u. 664) und eine eingefügt (ein T nach Nukleotid 916).
4.4.1 IR - 'Inverted Repeats'
Bei IR handelt es sich um funktionelle Bereiche von IS-Elementen, die die Bindestelle für die Transposase darstellen. Deletionsversuche bei IS911 haben gezeigt, daß für die Transposition die volle Länge des IR (38 bp) notwendig ist [Polard et al., 1991]. Ein Vergleich der IR von 'IS3Ea9' mit den IR anderer IS-Elemente der IS3-Familie ergab eine hohe Konservierung, wie aus Tabelle 10 deutlich hervorgeht. Der Vergleich von linken und rechten IR zeigt jedoch auch, daß es sich bei allen IS-Elementen um 'imperfekte' IR handelt. Die bei 'IS3Ea9' gefundenen IR zeigen 100 % Homologie zu dem für IS3-Elemente charakteristischen Consensus (Tab.10).
Tabelle 10: Terminale 'Inverted Repeats' von IS3-Elementen
_________________________________________________________________________
IS IR
_________________________________________________________________________
| IS150 | R | TGTACTGACCCCAAAAAGTTGGACAGTTAAACACGA | ||||||||||
| L | TGTACTGCACCCATTTTGTTGGACGATGAAATGGAA | |||||||||||
| IS861 | R | TGTACTGACCCCCAAAAGTTGGACAATTTATTTTAAG | ||||||||||
| L |
TGAACTGCACCCCAAAAGTTAGACAAAAAATTTAACG |
|||||||||||
| IS3411 | R | TGAACCGCCCCGGGTTTCCTGGAGAGTGTTTTATCTGTGA | ||||||||||
| L | TGAACCGCCCCGGGAATCCTGGAGACTAAACTTCCTGAGA | |||||||||||
|
IS911 |
R |
TGAAGTGGCACACTGAATTTGGCCACCTGAACAGAGGT |
||||||||||
|
L |
TGAAGTGGTCAACAAAAACTGGCCACCGAGTTAGAGTT |
|||||||||||
|
IS3 |
R |
TGATCCTACCCACGTAATATGGACACAGGCCTAAGCGAG |
||||||||||
|
L |
TGATCTTACCCAGCAATAGTGGACACGCGGCTAAGTGAG |
|||||||||||
|
IS476 |
R |
TGACCTGCCCCCATCGT-CCGTACCAGCTGAAGCTATAAA |
||||||||||
|
L |
TGACCTGCCCCCACTGAGCCGTACCAGTGATTACTGATAA |
|||||||||||
|
IS407 |
R |
TGACCTGCCCCCATCAATAGGGCCAATG-GGC-TCTAGCAAAGTCC |
||||||||||
|
L |
TGACCTGCCCCCTGCAAACAGGGCCAGCCGGAGTCTAGTAAAGTTC |
|||||||||||
|
ISR1 |
R |
TGACGTGACCCCCGTTT-CTGATCCAGCCATAACGAGAGTCC |
||||||||||
|
L |
TGACGTGACCCCCTGAAACTCCTCCAGGAATAGCTAGAGTCC |
|||||||||||
|
ISEa9 |
R |
TGACCTGCCCCCAG--GATTAGATACAACGCTCACTTAGTAATGTC |
||||||||||
|
L |
TGACCTGCTCCCAGTTGATTAG-TACACCCCGATGTTAGTAATGTC |
|||||||||||
|
Consensus |
+++ +++ ++++.......+++ + |
|||||||||||
_________________________________________________________________________
Tabelle 10: Die Referenzen zu den aufgeführten IS-Elementen sind in Tabelle 9 angegeben. L und R bezeichnen jeweils das rechte bzw. linke terminale IR des IS-Elementes; 'Mismatches' zwischen links und rechts wurden fett gedruckt. Bei allen aufgeführten IR konservierte Bereiche (identisch bei über 80 %) wurden im Consensus mit + markiert.
4.4.2 ORFA
Entsprechend einer inzwischen etablierten Konvention für die Benennung der offenen Leseraster bei IS3-Elementen (vgl. Einleitung, S.4-5) wird das kleine ORF am a-Terminus des IS-Elementes als orfA, das Genprodukt als ORFA bezeichnet. Da frühere Publikationen diese Vereinheitlichung noch nicht aufwiesen, erfolgte eine entsprechende Umbenennung. Die Definition, welches offene Leseraster eines IS-Elementes aus der Erstbeschreibung im folgenden als ORFA angesprochen wird, wurde in Tabelle 11 aufgeführt.
Für das IS-Element 'IS3Ea9' erstreckt sich orfA von Nukleotid 465 bis 725 und hat eine Größe von 87 aa.
Tabelle 11: Erläuterung zur Bezeichnung 'ORFA'
_________________________________________________________________________
|
Bezeichnung des ORFA in der Erstbeschreibung |
Größe [aa] |
_________________________________________________________________________
|
IS3 |
ORFII |
99 |
||
|
IS150 |
ORFA |
173 |
||
|
IS3411 |
ORFI |
108 |
||
|
IS861 |
ORF 1 |
141 |
||
|
ISR1 |
ORFA3 |
88 |
||
|
IS911 |
ORFA |
100 |
||
|
IS476 |
-1 |
88 |
||
|
IS407 |
ORF 4 |
88 |
||
|
'IS3Ea9' |
ORFA |
87 |
_________________________________________________________________________
Tabelle 11: Bei den mit 1 markierten IS-Elementen wurden dieser ORF noch nicht beschrieben. Nach Korrektur der Sequenz (vgl. Tab. 9 ) ergab sich das offene Leseraster der angegebenen Größe.
Aus Literaturangaben war bekannt, daß sich ORFA von IS-Elementen als DNA-bindendes Protein durch einen stark basischen Charakter auszeichnet, was sich in einem stark basischen isoelektrischen Punkt (pI) niederschlägt [Machida et al., 1982; Prentki et al., 1987a; Polard et al., 1991]. Das dem ORFA bei IS1 entsprechende Protein weist einen pI=10.9 auf [Galas u. Chandler, 1989].
Da in der jeweiligen Referenzliteratur konkrete Zahlenangaben für die IS3-Familie fehlten, wurde mit Hilfe des GCG-Programms ISOELECTRIC für die oben aufgeführten IS3-Elemente der pI von ORFA bestimmt und es zeigten sich vergleichbar basische Werte; beispielsweise hat ORFA von IS911 einen isoelektrischen Punkt von pI=10.6.
Die Untersuchung der Zusammensetzung des ORFA von 'IS3Ea9' wurde ebenfalls mit Hilfe des GCG-Programms ISOELECTRIC durchgeführt. Das Ergebnis ist in Abbildung 19 dargestellt.
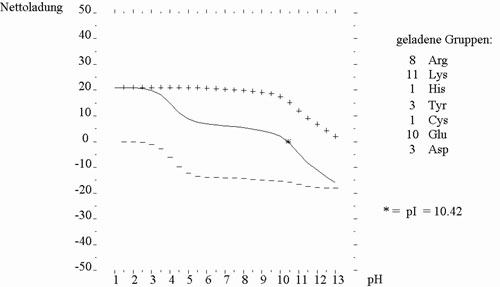
Es zeigte sich auch hier ein basischer isoelektrischer Punkt von pI=10.4, was auf eine positve Nettoladung in einem neutralen (pH 7) Milieu hinweist und ebenfalls DNA-bindende Eigenschaften für ORFA nahelegt. Vergleichbare Zahlenwerte finden sich, abgesehen von IS-kodierten Proteinen, bei Prokaryonten sonst nur bei histonartigen Proteinen (wie HU) oder IHF, deren DNA-Bindungseigenschaften nachgewiesen sind.
Abbildung 19: Abgeleitete Titrationkurve des ORFA von 'IS3Ea9'
_________________________________________________________________________
_________________________________________________________________________
Abbildung 19: Die Nettoladung von ORFA ist gegen den pH-Wert in Form einer Titrationskurve aufgetragen. Der isoelektrische Punkt wurde mit * gekennzeichnet. Die von der AS-Sequenz abgeleitete Analyse wurde mit Hilfe des GCG-Programms ISOELECTRIC durchgeführt.
Um konkrete Homologien auf AS-Ebene mit dem ORFA anderer IS-Elemente zu finden wurde ein multiples Alignment mit dem jeweiligen ORFA von IS407, IS476, ISR1, IS3, IS3411, IS911, IS861 und IS150 unter Verwendung des HUSAR-Programms TREE durchgeführt. Der Algorithmus dieses Programm berücksichtigt im Vergleich zu herkömmlichen Programmen zum Alignment von DNA- und AS-Sequenzen verstärkt Evolutionsprinzipien, wie z.B. den Grundsatz 'einmal eine Lücke, immer eine Lücke', was für eine spätere Stammbaumerstellung von Bedeutung ist [Feng u. Doolittle, 1987]. Das resultierende Alignment ist in Abbildung 20 dargestellt. Der Vergleich der AS-Sequenzen von ORFA zeigt in großen Bereichen eine hohe Konservierung, was auf das Vorhandensein von funktionell wichtigen Domänen schließen läßt. Insgesamt beträgt die Sequenzhomologie bei den aufgeführten IS-Elementen auf AS-Ebene jedoch nur 30-65 %. Bereits der visuelle Vergleich der AS-Sequenzen von ORFA zeigt, daß sich innerhalb der IS3-Familie Untergruppen hoher Sequenzhomologie bilden.
Abbildung 20: Multiples Alignment von ORFA
_________________________________________________________________________
10 20 30 40 50 60 70
---------.---------.---------.---------.---------.---------.---------.
IS407 MKK R FTEQQIIGFLKEA EAGM
'IS3Ea9' MKK R FSDQQIISILREA EAGV
IS476 MKK SR FSTEQIIGFIKQA DAGM
ISR1 MKR SR FTEEQIIGILREQ EAGV
IS3 MTKTVSTSKK PRKQHSPEFRSEALKLAERI
IS3411 MTKN TR FSPEVRQRAVRMVLESQSEYDSQWA
IS911 MKK RN FSAEFKRESAQLVVD QNY
IS861 MIK LMDR YGVEIVEKGRNEYYPPELKQEMIDK
IS150 MSKPKYPFEKRLEVVNHYFTTDDGYRIISARFGVPRTQVRTWVALYEKHGEKGLIPKPKGVSADPELRIK
+++ + ++ ++++ ++++ ++++
80 90 100 110 120 130 140
---------.---------.---------.---------.---------.---------.---------.
IS407 PVKELCRKH GFSD ASFYTWRAKF GGMEVSEARR LKDLE
'IS3Ea9' SARELCRKH AISD ATFYTWRKKY GGMEVPEVKR LKSLE
IS476 AVAELCRRH GFSP ASFYQWRAKY GGMEADEAKR LKELE
ISR1 ATAEVCRRH GVSS ATFYKWKAKF GGLDVSEARR LKALE
IS3 GVTAAAREL SLYE SQLYNWRSKQ QNQQTSSERE L EMS
IS3411 TICSIAPKI GCTR ETLRVWVRQ HERDTGGGD GGLTT
IS911 TVADAAKAM DVGL STMTRWVKQL RDERQGKTPK ASPIT
IS861 VLIHGCSQL SVSLDYALSNCSILTNWLSQFKKNGYTIVEKTRGRPSKMGRKRKKTWEEMTELERLQ
IS150 VVKAVIEQHMSLNQAAAHFMLAGSGSVARWLKVYEERGEAGLRALKIG TKRNIAISVD
++++++ + + ++ +++++ ++++++ ++ ++ ++
150 160 170 180 185
---------.---------.---------.---------.-----
IS407 VENARLKK L LAEAMLDMEALKVV VKG KP
'IS3Ea9' EENARLKK L LAEAMLDKEALQVA LGR KY
IS476 VQNTRLKK L LAEAHLDIEALKVG FGV KR
ISR1 DENARLKR M LADAMLDNVALKDL LGK KW
IS3 TEIARLKR Q LAERDEELAILQKAATYFAK RLK
IS3411 AERQRLKE LERENRELRRSNDILRQASAYFAK AEFDRLWKK
IS911 PEQIEIRE LRKKLQRIEMENEILKKATALLMS DSLNSSR
IS861 EENERLRTENAFLKKLRDLRLRDEALQSERQKQLE KWSQEDSD
IS150 PEKAASALELSKDRRIEDLERQVRFLETRLMYLKKLKALAHPTKK
+++++++ + ++++ ++ ++ + +
_________________________________________________________________________
Abbildung 20: Die AS-Sequenz von ORFA ist für einige IS-Elementer der IS3-Familie in einem multiplen Alignment dargestellt. Konservierte Bereiche sind mit + markiert (konserviert bei mind. 50% aller aufgeführten IS-Elemente). Referenzen sind in Tabelle angegeben. Das Alignment wurde mit Hilfe des HUSAR-Programms TREE [Feng u. Doolittle, 1987] erstellt.
In der Referenzliteratur für die genannten IS-Elemente wird als ein funktioneller Bereich ein Helix-Turn-Helix-Motiv im ORFA beschrieben, ein charakeristisches Merkmal für Proteine mit DNA-Bindungseigenschaften. Um festzustellen, ob sich im ORFA von 'IS3Ea9' ebenfalls ein solches Motiv befindet, wurde die AS-Folge von ORFA anhand von Kalkulationstabellen [Dodd u. Egan, 1987], wie unter 3.9.4 beschrieben, untersucht. Ein Vergleich der postulierten Helix-Turn-Helix-Motive ist in Tabelle 12 aufgeführt.
Tabelle 12: Potentielle Helix-Turn-Helix-Motive im ORFA bei IS3-Elementen
_________________________________________________________________________
|
Pos. im ORFA |
Helix |
Turn |
Helix |
Score |
|||||||||||
|
1ISR1 |
(24) |
TAEVCRRH |
GVS |
SATFYKWKA |
1961 |
||||||||||
|
1 IS3411 |
(33) |
ICSIAPKI |
GCT |
RETLRVWVR |
1757 |
||||||||||
|
1 IS911 |
(25) |
VADAAKAM |
DVG |
LSTMTRWVK |
1464 |
||||||||||
|
1 IS476 |
(24) |
VAELCRRH |
GFS |
PASFYQWRA |
1315 |
||||||||||
|
'IS3Ea9' |
(23) |
ARELCRKH |
AIS |
DATFYTWRK |
1230 |
||||||||||
|
IS407 |
(23) |
VKELCRKH |
GFS |
DASFYTWRA |
1131 |
||||||||||
|
1 IS150 |
(25) |
YRIISARF |
GVP |
RTQVRTWVA |
1051 |
||||||||||
|
1 IS3 |
(32) |
VTAAAREL |
SLY |
ESELYNWRS |
856 |
||||||||||
|
1 IS861 |
(27) |
QEMIDKVL |
IHG |
CSQLSVSLD |
782 |
||||||||||
_________________________________________________________________________
Tabelle 12: Links wurden IS-Elemente der IS3-Familie, daneben mögliche Helix-Turn-Helix-Motive aufgeführt; die Zahlenangabe in Klammern gibt die Position der ersten AS des Motiv im ORFA an. Bei mit 1 gekennzeichneten IS-Elementen wurden Angaben von Prère übernommen [Prère et al., 1990]. Die IS-Elemente wurden nach ihrem Score-Wert angeordnet. Der Score-Wert für das Motiv wurde mit der Dodd-Egan-Wichtungsmatrix berechnet [Dodd u. Egan, 1987].
Der resultierende 'Score'-Wert bestätigte im Vergleich mit anderen IS-Elementen (Tab.12) das Vorhandensein der gesuchten Sekundärstruktur, wonach sich demnach auch im ORFA von 'IS3Ea9' von Aminosäure 23 bis 43 mit hoher Wahrscheinlichkeit ein Helix-Turn-Helix-Motiv befindet.
4.4.3 ORFB
Analog den Verhältnissen zur Nomenklatur bei ORFA von IS3-Elementen, wird das große offene Leseraster am ß-Terminus des IS-Elementes, welches in Relation zu orfA in Phase -1 kodiert, als orfB und das Genprodukt als ORFB bezeichnet. Die Definition, welches offene Leseraster aus der Erstbeschreibung im folgenden als ORFB angesprochen wird, wurde in Tabelle 13 aufgeführt. Das ORFB von 'IS3Ea9' erstreckt sich von Nukleotid 754 bis 1582 und kodiert für 276 aa.
Tabelle 13: Erläuterung zur Bezeichnung 'ORFB'
_________________________________________________________________________
Bezeichnung des ORFB in der Erstbeschreibung |
Größe [aa] |
_________________________________________________________________________
|
IS3 |
ORFI |
288 |
||
|
IS150 |
ORFB |
283 |
||
|
IS3411 |
ORFV |
282 |
||
|
IS861 |
ORF 2 |
277 |
||
|
ISR1 |
-1 |
305 |
||
|
IS911 |
ORFB |
272 |
||
|
IS476 |
-1 |
277 |
||
|
IS407 |
ORF 1 |
277 |
||
|
'IS3Ea9' |
ORFB |
276 |
_________________________________________________________________________
Tabelle 13: Bei mit 1 gekennzeichneten IS-Elementen wurde dieser ORF in der Erstpublikation noch nicht beschrieben. Nach Korrektur der Sequenz (vgl. Tab.9) ergab sich ein offenes Leseraster der angegebenen Größe.
Auch für ORFB war nach Literaturangaben ein basischer Charakter zu erwarten. Da auch hier, wie bei ORFA, konkrete Zahlenwerte zum Vergleich mit ORFB von 'IS3Ea9' fehlten, wurde zunächst für die ORFB-Proteine verschiedener IS3-Elemente der isoelektrische Punkt mit Hilfe des GCG-Programms ISOELECTRIC bestimmt. Es zeigte sich in allen Fällen ein dem ORFA vergleichbar basischer pI; für ORFB von IS911 wurde beispielsweise ein isoelektrischer Punkt von pI=10.8 ermittelt.
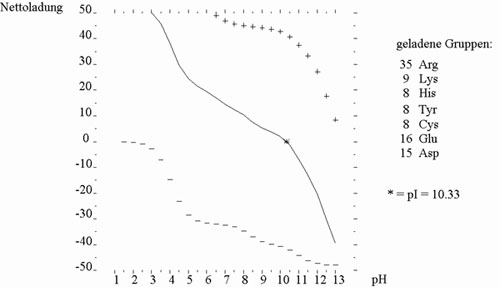
Zur Abschätzung der Eigenschaften des ORFB von 'IS3Ea9' wurde ebenfalls das GCG-Programm ISOELECTRIC verwendet. Das Ergebnis ist in Abbildung 21 dargestellt. Vergleichbar mit ORFA hat auch ORFB einen auffällig basischen pI von 10.3.
Abbildung 21: Abgeleitete Titrationskurve des ORFB von 'IS3Ea9'
_________________________________________________________________________
_________________________________________________________________________
Abbildung 21: Die Nettoladung von ORFB ist in Abhängigkeit vom pH-Wert in Form einer Titrationskurve aufgetragen. Der isoelektrische Punkt wurde mit * gekennzeichnet. Die von der AS-Sequenz abgeleitete Analyse wurde mit Hilfe des GCG-Programms ISOELECTRIC durchgeführt.
Um festzustellen, ob ORFB von 'IS3Ea9' ein hohes Maß an Homolgie zum ORFB anderer IS3-Elemente zeigt, wurden die jeweiligen AS-Sequenzen von IS407, IS476, ISR1, IS3, IS3411, IS911, IS861, IS150 und 'IS3Ea9' mit Hilfe des HUSAR-Programms TREE [vgl. Anmerkung unter 4.4.2; Feng u. Doolittle, 1987] in einem multiplen Alignment zusammengestellt (Abb.22).
Abbildung 22: Multiples Alignment von ORFB
__________________________________________________________________________
10 20 30 40 50 60 70
---------.---------.---------.---------.---------.---------.---------.
IS476 ML EHTPLSERRACRLAGLSRDAFRH APVPTPATQALSARLVEL
'IS3Ea9' MLIC DATGLSQRRACRLTGLSLSTCRY EAQRPAADAHLSGRITEL
IS407 MI
ISR1 VVTPAGYREAAGHLQAAYEMSERRACRVLGVDRTSVRY QATRPDDASARPA EGP
IS150 VKVLN ELRQFYPLDELLRAAEIPRSTFYYHLKALSKPDKYADVKKRISEI Y
IS861 MVTG GFR LDLLLEITKIARATYYYQLKKLNKPDKDKAIKSDIQSI Y
IS3 MKYVFIE KHQAEFSIKAMCRVLRVARSGWYTWCQRRTRISTRQQFRQHCDSV VLAAF
IS911 VVTLCHVFGVHRSSYRYWKNRPEKPDGRRAV LRSQVL ELHGI
IS3411 MMPLLD KLREQYGVGPLCSELHIAPSTYYHCQQQRHHPDKRSARAQRDDWLKKQIQRVY
+ +
80 90 100 110 120 130 140
---------.---------.---------.---------.---------.---------.---------.
IS476 AQTHRRFGYRRLHDLLRPEFPSVNHKKIYRLYEEAELKVRKRRKA KRPVGE RQKLLAS
'IS3Ea9' ALERRRFGYRRIWQLLRREGLHVNHKRVYRLYHLSGLGVKRRRRR KGLATE RLPLLRP
IS407 E REQLALP
ISR1 GQERRRFGYRRLHVLLRREGHAVNRKRVQRIYRDEQLTV RRRA PQASDG HAATVTC
IS150 HENRGRYGYRRVTLSLHREGKQINHKAVQRLMGTLSLKAAIKVKR YR SYRGEVGQTAPNVLQRDFKA
IS861 DEHRGNYGYRRIYLELRNRGFVINHKRVQGLMKSMGLTARIRRKRKYA SYKGEVGKKADNLIQRQFEG
IS3 TRSKQRYGAPRLTDELRAQGYPFNVKTVAASLRRQGLRA KASRKFSPVSYRAHGLPVSENLLEQDFYA
IS911 SHGSA GARSIATMATRRGYQMGRWLAGRLMKELGLVSCQQPTHRYKRGGHEHVAIP NYLERQFAV
IS3411 DENHKVYGVRKVWRQLLREGIRVARCTVARLMAVMGLAGVLRGKKVRTTISRKAVAAG HRVNRQFVA
+++++++++ +++++ +++ +++++ ++ ++++ +++ + + ++
150 160 170 180 190 200 210
---------.---------.---------.---------.---------.---------.---------.
IS476 SMPNDTWSMDFVFDALANARRIKCLTVVDDFTRESVDIAVDHGISGAYVVRLLDQA ACFRGYPRAVR
'ISEa9' AAPNLTWSDDFVMDALATGRRIKCLTCVDDFTKECLTVTVAFGISGVQVTRILDSI ALFRGYPATIR
IS407 GAPNEVWSIDFVMDALSNGRRVKCLTVVDDFTKEAVDIVVDHGISGLYVARALDRA ARFRGYPKAVR
ISR1 RWRQPALVADFVADQLTDGRRFRILTVIDNCTRECLALVADTSLSGARVVRELDAV IRQRGRPDTIV
IS150 TRPNEKWVTDVTEFAVNGRKLYLSP VIDLFNNEVISYSLSERPVMNMVENMLDQAFK KLNPHEHPVLH
IS861 SKPYEKCYTDVTEFALPEGKLYLSP VLDGYNSEIIDFTLSRSPALKQVQTMLERAFP A ASYSETILH
IS3 SGPNQKWAGDITYLRTDEGWLYLAV VIDLWSRAVIGWSMSPRMTAQLACDALQMALW RRKRPRNVIVH
.IS911 TEPNQVWCGDVTYIWTGKRWAYLAV VLDLFARKPVGWAMSFSPDSRLTMKALEMA WETRGKPGGVMFH
IS3411 ERPDQLWVADFTYVSTWRGFVYVAF IIDVFAGYIVGWRVSSSMETTFVLDALEQALW TRSTAGTVHH
++ + ++++++ +++++ + + ++++++ + + + + + + +++
220 230 240 250 260 270 280
---------.---------.---------.---------.---------.---------.---------.
IS476 TDNGPEFTSRAFIAWTQQHGIEHILIEPGAPTQNAYIESFNGKFRDECLNEHW FTSLAQ ARDVIAD
'ISEa9' TDQGPEFTCRALDQWAFEHGVELRLIQPGKPTQNGFIESFNGRFRDECLNEHW FSDVSH ARKTISE
IS407 TDQGPEFTSRALDQWAYANGVTLKLIQAGKPTQNAYIESFNGKFRDECLNEHW FTTLAH ARAVIAA
ISR1 SDNGTEYTSNAVLAWATTPAFGWHYIAPGKPQQNGFNESFNGRLRDELLNETL FRSLPPLAPCGGVAT
IS150 SDQGWQYRMRRYQNILKEHGIKQSMSAKGNCLDNAVVECFFGTLKSECFY LD EFSNISE LKDAVTE
IS861 SDQGWQYQHKSYHQFLEDKGIRPSMSRKGNSPDNGMMESFFGILKSEMFYGLEKSYKSLDD LEQAITD
IS3 TDRGGQYCSADYQAQLKRHNLRGSMSAKGCCYDNACVESFFHSLKVECIHGEH FISREI MRATVFN
IS911 SDQGSHYTSRQFRQLLWRYQIRQSMSRRGNCWDNSPMERFFRSLKNEWIPVVG YVSFSE AAHAITD
IS3411 SDKGSQYVSLAYTQRLKEAGLLASTGSTGDSYDNAMAESINGLYKAEVIHRKS WKNRAE VELATLT
++++ ++ ++ ++ ++ +++++ + ++ + +
290 300 310 320 330 340
---------.---------.---------.---------.---------.---------.-
IS476 WRRH YN QIRP HSSCGRIPPA QFAANYRTQQANNAVPFNPGLYQ
'ISEa9' WRQD YN EYRP HSALNYQAPS EFAAAWRKGNSDSE GSDITK
IS407 WRQD YN EQRP HSALNYLAPS EFAAKHRATADAPAA FQELV
ISR1 RLQR AASALEARVADAAGLCRSAHRTQRPACCVVDGCADRPLANPADHSSDQPRTLVMAG
IS150 YIEY YN SRRI SLKLKGLTPI EYRNQTYMPRV
IS861 YIFY YN NKRI KAKLKGLSPV QYRTKSFT
IS3 YIECDYN RWRR HSWCGGLSPE QFENKNLA
IS911 YIVGYYS ALRP HEYNGGLPPN ESENRYWKKL
IS3411 WVDW YN NRRL LERLGHTPPA EAE
++++ ++ ++ ++ + + +++
__________________________________________________________________________
Abbildung 22: Die AS-Sequenzen des ORFB von IS-Elementen der IS3-Familie sind hier in einem multiplen Alignment dargestellt. Konservierte Bereiche sind mit + markiert (konserviert bei mind. 60 % aller aufgeführten IS-Elemente). Referenzen sind in Tabelle 9 aufgeführt. Das Alignment wurde mit Hilfe des HUSAR-Programms TREE [Feng u. Doolittle, 1987] erstellt.
Vergleichbar mit ORFA zeigten sich auch bei ORFB, mit Ausnahme des Bereiches der ersten 70 AS, Regionen hoher Konservierung auf AS-Ebene, was auf das Vorhandensein funktionell wichtiger und insofern konservierter Domänen schließen läßt.
In der Literatur werden an dieser Stelle des öfteren Homologievergleiche von IS-kodierten mit retroviralen Genen aufgeführt [Chandler u. Fayet, 1993]. Konkret wurden Homologien zu einer funktionellen Domäne der Intergrase von Retroviren für IS911 beschrieben [Khan et al., 1991].
Datenbankrecherchen mit ORFB von 'IS3Ea9' ergaben zunächst kein eindeutiges Ergebnis. Erst nach gezieltem Alignment des ORFB von 'IS3Ea9' mit der Sequenz der Integrase eines endogenen Retrovirus der Maus (MIn) wurde in einem Bereich von 74 aa eine mäßige Übereinstimmung von 23 % Identität festgestellt (Abb.23). ORFB von IS911 zeigte beim Alignment mit der MIn-Sequenz eine vergleichbar geringe Homologie (Ergebnis nicht abgebildet).
Abbildung 23: Alignment von 'IS3Ea9' ORFB und MIn
_________________________________________________________________________
|
30 40 50 60 70 80 |
|
|
'IS3Ea9' |
LSTCRYEAQRPAADAHLSGRITELALERRRFGYRRIWQLLRREGLHVNHKRVYRLYHLSG |
|
|: : |:| | :| : :: | : : : |
|
|
MIn |
XXAAARGFQTSRCTAWRACKXAWREGLLVNFDISGSWKLTRGGG--APYRISYGVSPWGP |
|
100 110 120 130 140 150 |
|
|
90 100 110 120 130 140 |
|
|
'IS3Ea9' |
LGVKRRRRRKGLATERLPLLRPAAPNLTWSDDFVMDALATGRRIKCLTCVDDFTKECLTV |
|
:: |:| || : : :|:: |: ||: : :|: |:|:: : |
|
|
MIn |
GVLRARXRAKG-SRSTQSLVPVASVNLVKVSLIVLFILSTXPELWGLXAQCSFQSIPSSL |
|
160 170 180 190 200 210 |
_________________________________________________________________________
Abbildung 23: Alignment des ORFB von 'IS3Ea9' und MIn - Integrasedomäne eines endogenen Retrovirus der Maus (Khan et al., 1991). Identische AS sind in der Gegenüberstellung beider Sequenzen mit einer Linie, ähnliche AS mit zwei Punkten verbunden.
Über die Funktion und Eigenschaften von ORFB ist wenig bekannt. Lediglich die basische Nettoladung des Proteins deutet auf DNA-Bindungseigenschaften. Ausgehend von Sequenzhomologien im ORFB zur Integrase von Retroviren ließe sich über funktionelle Gemeinsamkeiten spekulieren, schließlich verfügt das retrovirale Protein über die Funktion der Incision, Integration und Religation von DNA - alles Eigenschaften, die auch für die Transposase von IS-Elementen gefordert werden [Fayet et al., 1990; Khan et al., 1991; Polard et al., 1991].
4.4.4 ORFC
Das gefundene offene Leseraster orfC liegt im Bereich von Position 988-1230 auf dem Gegenstrang zu orfA und orfB. Eine 'upstream' liegende Promotorstruktur mit Homologie zum s70-Promotor von E.coli wurde zwischen den Nukleotiden 1269 und 1308 beschrieben, jedoch zeigte die vermutete Shine-Dalgarno-Sequenz nur eine geringe Homologie zur SD-Consensus-Sequenz. Eine Translation von orfC wäre demnach unwahrscheinlich.
Ein Vergleich mit IS-Elementen der IS3-Familie zeigte, daß das jeweilige Leseraster auf dem Gegenstrang, relativ zu orfA und orfB, ohne weitere Bedeutung für die Funktion und Effizienz der Transposition ist. Da auch innerhalb dieser IS-Gruppe keine Konservierung in Bezug auf Größe und Lage festzustellen war, wurde orfC im weiteren nicht näher charakterisiert.
4.4.5 Motive für ribosomales 'Frameshifting'
Die Gruppe der IS3-Elemente zeichnet sich neben ihrer weiten Verbreitung nicht allein durch die Struktur der offenen Leseraster orfA und orfB in Phase 0 und -1, sondern auch durch Übereinstimmungen in vielen charakteristischen Merkmalen mit einer weiteren großen Gruppe transponierbarer Elemente aus - den Retroviren [Fayet et al., 1990]. Die Sequenz der IR ist den retroviralen LTRs (Long Terminal Repeats) äußerst ähnlich. Insbesondere die terminalen Dinukleotide 5'-TG...CA-3' sind bei IS3-Elementen und Retroviren zu 100 % konserviert [Skalka, 1988]. Desweiteren zeigt der ORFB der IS3-Elemente, wie bereits unter 4.4.3 erwähnt, Homologien zu einer hochkonservierten Region in retroviralen Integraseproteinen [Fayet et al., 1990; Khan et al., 1991]. Inzwischen wird eine weitere Übereinstimmung beider Gruppen postuliert, wonach IS3-Elemente ihre Transposase durch einen Mechanismus bilden, der ein ribosomales 'Frameshifting' von Phase 0 nach -1 beinhaltet [Polard et al., 1991].
Nachgewiesen war dieser Mechanismus für die gag-pol-Gene vieler Retroviren [Jacks et al., 1988; Weiss et al., 1990]. Inzwischen wurde dieses Prinzip jedoch auch für IS-Elemente belegt: Bei IS1 bilden insA und insB ein InsAB-Fusionsprotein mittels ribosomalem 'Frameshifting' [Sekine u. Ohtsubo, 1989]. Bei IS3-Elementen erfolgte der Nachweis des 'Frameshifting' für IS911 [Polard et al., 1991] und IS150 [Vögele et al., 1991]. In diesem Zusammenhang wurden zwei strukturelle Mechanismen, sogenannte 'Frameshift-Fenster', postuliert: Typ I beinhaltet ein Hepta-nukleotid-'Frameshift-Fenster' der allgemeinen Form Y YYX XXZ (Phase 0), bzw. YYY XXX Z (Phase -1), das am häufigsten anzutreffen ist. Die Sequenz A AAA AAG, die bei vielen Retroviren und IS-Elementen, aber auch im dnaX-Gen von E.coli vorzufinden ist, bildet dabei das effizienteste 'Frameshift-Fenster' mit einer 'Frameshifting'-Frequenz von ca. 90 % [Atkins, zitiert in Chandler u. Fayet, 1993]. Ein wesentlicher Anteil an dieser hohen Effizienz kommt dabei dem AAG-Codon zu, da E.coli das entsprechende CUU-tRNA-Anticodon fehlt [Tsuchihashi u. Brown, 1992]. 'Frameshifting' des Ribosoms nach Typ II mit einer vielfach reduzierten Effizienz beinhaltet ein kurzes Sequenzmotiv meist vier identischer Basen der allgemeinen Form X XXX oder X XXZ (Phase 0), bzw. XXX Z (Phase -1). Notwendige Voraussetzung ist allerdings ein kurz darauf folgendes Stoppcodon [Weiss et al., 1990]. Trotz der dabei entstehenden Fehlpaarungen bei der Codon-Anticodon-Paarung der tRNA konnte ribosomales 'Frameshifting' im Gen10 des Phagen T7 nachgewiesen werden [Condron, zitiert in Chandler u. Fayet, 1993].
Potentielle 'Frameshift-Fenster' vom Typ I und -II sind für einige IS-Elemente in Tabelle 14 aufgelistet.
Tabelle 14: Potentielle 'Frameshift-Fenster' bei IS-Elementen
_________________________________________________________________________
|
IS |
'Frameshift-Fenster' |
_________________________________________________________________________
|
1 IS1 |
T GAT-20- AAC ACG ATT TTC CGC CAT TTA AAA AAC TCA -4-TAA |
||
|
1 IS150 |
T GAA-6- GAA ACG CGG CTT ATG TAT CTA AAA AAG CTG -9-TAG |
||
|
IS861 |
T GAG-6- AGA ACG GAG AAT GCC TTT CTA AAA AAG TTG -26-TAG |
||
|
1 IS911 |
ATG GAG AAT GAA ATA TTA AAA AAG GCT -12-TAA |
||
|
IS407 |
T GAC-43- GAA GCG TTG GTC GTG GTC AAG GGA AAG CCC TGA |
||
|
ISR1 |
T GAG-16- GCC CTG AAG GAC CTG CTG GGA AAA AAG TGG TGA |
||
|
IS3 |
T GAA GAG CTG GCT ATC CTC CAA AAG GCC -22-TGA |
||
|
IS3411 |
T GAC CGC CTC TGG AAA AAA TGA |
||
|
IS476 |
GTT GGC TTC GGG GTA AAA CGC TAG |
||
|
'IS3Ea9' |
T AAA GAG GCG CTT CAG GTG GCT CTT GGG CGA AAG TAC TGA |
_________________________________________________________________________
Tabelle 14: Typ I- Heptanukleotide (oben) und mögliche Typ II -'Frameshift'-Motive (unten) sind fett-gedruckt und unterstrichen. IS-Elemente, bei denen ribosomales 'Frameshifting' nachgewiesen wurde, sind mit 1 markiert [Chandler u. Fayet, 1993].
Eine Modulation der 'Frameshift'-Effizienz wurde für verschiedene weitere Faktoren belegt. So bewirken Sekundärstrukturen der mRNA, wie Haarnadelschleifen [Jacks et al., 1988] oder Pseudoknoten, d.h., weitere Faltungen der Haarnadelschleifen, eine erhöhte 'Frameshift'-Frequenz [Tu, zitiert in Chandler u. Fayet, 1993; Tzy-Hwa et al., 1992]. Die gleiche Wirkung haben 'upstream' gelegene Ribosomen-Bindestellen vom Typ der Shine-Dalgarno-Sequenz [Prère, zitiert in Polard et al., 1991]. Als allgemein gültiges Prinzip ließe sich formulieren, daß sämtliche Faktoren, die die Wanderungsgeschwindigkeit des Ribosoms auf der mRNA herabsetzen, gleichzeitig die 'Frameshifting'-Frequenz erhöhen [Chandler u. Fayet, 1993]. Dementsprechend konnte gezeigt werden, daß die Bindung spezifischer Proteine an die mRNA ebenso wie die Verwendung seltener Codons das 'Frameshifting' stimuliert [Dix, 1990].
Von den 23 zur Zeit bekannten Vertretern der IS3-Familie weisen 11 IS-Elemente ein Typ I -Heptanukleotid-'Frameshift-Fenster' auf. Bei IS3, IS3411 und IS476 liegt ein potentielles Typ II -Motiv vor, wobei IS3411 dieses Motiv gleich zweimal aufweist.
In 'IS3Ea9' befindet sich am 3'-Ende des orfA das Sequenzmotiv A AAG -3-TGA, begleitet von einem 'Inverted Repeat', das auf mRNA-Ebene zur Ausbildung von haarnadelschleifen-förmigen Sekundärstrukturen führen könnte. Unter Verwendung des GCG-Programms FOLD/SQUIGGLES wurde für die Sequenzbereiche des möglichen 'Frameshift-Fensters' von 'IS3Ea9' und des bereits nachgewiesenen Fenster von IS911 die mögliche Faltung einer mRNA bestimmt. Für 'IS3Ea9' ist das Ergebnis in Abbildung 24 dargestellt. Die mRNA-Faltung für IS911 zeigte eine zu 'IS3Ea9' vergleichbare Haarnadelschleifenform (nicht abgebildet), die auch den in der Literatur angegeben Strukturen entsprach [Chandler u. Fayet, 1993].
Abbildung 24: Eine der möglichen mRNA-Strukturen im Bereich des 'IS3Ea9'-Frameshift-Fensters
_________________________________________________________________________
_________________________________________________________________________
Abbildung 24: Mit Hilfe des GCG-Programms FOLD/SQUIGGLES wurde für den Übergangsbereich von orfA-orfB eine der möglichen Sekundärstrukturen der mRNA konstruiert. UGA (fett gedruckt) kennzeichnet das Stoppcodon von orfA. AUG markiert das Startcodon und SD die Shine-Dalgarno-Sequenz von ORFB. Das potentielle Frameshift-Fenster beginnt bei Nukleotid 719 und ist durch fett gedruckte Linien hervorgehoben.
An der mRNA-Struktur im Bereich des 'Frameshift-Fensters' ist bemerkenswert, daß sämtliche für ein 'Frameshifting' notwendige Faktoren, wie (1) das 'Frameshift-Fenster', (2) das UGA-Stoppcodon und (3) die Shine-Dalgarno-Sequenz in schlecht oder ungepaarten Bereichen, d.h., an exponierter Stelle, vorkommen. Die UGA-Sequenz liegt wie bei IS150 und IS911 in der großen Schleife der Haarnadelstruktur [Polard et al., 1991; Chandler u. Fayet, 1993].
Da orfA und orfB jeweils in Phase 0 bzw -1 kodieren, die Position eines potentiellen 'Frameshift-Fensters' festgestellt wurde und insgesamt eine hohe Homologie zu IS3-Elementen nachgewiesen wurde, bestand die Möglichkeit, daß auch bei 'IS3Ea9' durch ribosomales 'Frameshifting' ein ORFAB-Fusionsprotein gebildet wird, das, wie für IS911 nachgewiesen, die aktive Transposase darstellt [Prère et al., 1990; Chandler u. Fayet, 1993]. Handelt es sich bei der Region zwischen orfA und orfB um eine intergenische Region im klassischen Sinne, so wird dieser Bereich nicht transkribiert und infolgedessen auch keiner Selektion unterworfen. Ein Vergleich dieser Übergangsbereiche zwischen orfA und orfB von verschiedenen IS3-Elementen dürfte demnach keine konservierten Bereiche aufweisen. Um dies im vorliegenden Fall zu überprüfen, wurden potentielle ORFAB-Fusionsproteine in einem multiplen Alignment verglichen, wobei ein Rasterschub am Ende von orfA von Phase 0 nach -1 am angegebenen 'Frameshift-Fenster' angenommen wurde (vgl. Tab.14). Das Alignment wurde mit Hilfe des HUSAR-Programms TREE [vgl. Anmerkung unter 4.4.2; Feng u. Doolittle, 1987] erstellt und ist unter Abbildung 25 aufgeführt. Der jeweilige orfA-B-Übergangsbereich wurde hervorgehoben.
Abbildung 25: Multiples Alignment potentieller ORFAB Fusionsproteine
_________________________________________________________________________
10 20 30 40 50 60 70
---------.---------.---------.---------.---------.---------.---------.
IS407 MKK RFTEQQIIG
'IS3Ea9' MKK RFSDQQIIS
IS476 MKK SRFSTEQIIG
ISR1 MKR SRFTEEQIIG
IS150 MSKPKYPFEKRLEVVNHYFTTDDGYRIISARFGVPRTQVRTWVALYEKHGEKGLIPKPKGVSADPELRIK
IS861 MIK LMDRYGV EIVEKGRNEYYPPELKQE
IS3 MTK TVSTSKKPRKQHSPEFRSE
IS911 MKK RNFSAEFKRE
IS3411 MTK NTRFSPEVRQRAVRMVLE
80 90 100 110 120 130 140
---------.---------.---------.---------.---------.---------.---------.
IS407 FLKE AEAGMPVKELCRKHGFSDAS FYTWR AKFGGMEVSEARRLKDLEVENA
'IS3Ea9' ILRE AEAGVSARELCRKHAISDAT FYTWR KKYGGMEVPEVKRLKSLEEENA
IS476 FIKQ ADAGMAVAELCRRHGFSPAS FYQWR AKYGGMEADEAKRLKELEVQNT
ISR1 ILRE QEAGVATAEVCRRHGVSSAT FYKWK AKFGGLDVSEARRLKALEDENA
IS150 VVKAVIEQHMSLNQAAAHFMLAGSGSVARWLKVYEERGEAGLRALKIGTKRNIAISVDPEKAASAL ELS
IS861 MIDKVLIHGCSQLSVSLDYALSNCSILTNWLSQFK KNGYTIVEKTRGRPSKMGRKRKKTWEEMTELE
IS3 A LKLAERIGVTAAARELSLYESQ LYNW RSKQQNQQTSSERELEMSTEIA
IS911 SAQLVVDQNYTVADAAKAMDVGLST MTRWVKQL RDERQGKTPKASPITPEQIEIR
IS3411 SQSEYDSQWATICSIAPKIG CTRETLRVWVR QHERDTGGGDGGLTTAERQRLK
150 160 170 180 190 200 210
---------.---------.---------.---------.---------.---------.---------.
IS407 RLKKLLAEAMLDMEALKVVVK GKALSPQAKREAVSAIRE KVNISERRACRLVGLSRSVLHY DAK
'IS3Ea9' RLKKLLAEAMLDKEALQVALG RKVLTTDQKREAVMLICD ATGLSQRRACRLTGLSLSTCRY EAQ
IS476 RLKKLLAEAHLDIEALKVGFG VKTLAPQRKREAIRRMLE HTPLSERRACRLAGLSRDAFRH APV
ISR1 RLKRMLADAMLDNVALKDLLG KKVVTPAGYREAAGHLQA AYEMSERRACRVLGVDRTSVRY QAT
IS150 K DRRIEDLERQVRFLETRLMYLKKAESLSSSHEKMKVLNELRQFYPLDELLRAAEIPRSTFYYHLKALS
IS861 RLQEENERLRTENAFLKKVEGFALEGRSL TERTAETIREMVTGGFRLDLLLEITKIARATYYYQLKKLN
IS3 RLKRQLAERDEELAILQKGRDILRE APEMKYVFIEKHQAEFSIKAMCRVLRVARSGWYTWCQRRT
IS911 ELRKKLQRIEMENEILKKGYRALDV RLPEQFSIIGKLRAHYPMVTLCHVFGVHRSSYRYWKNRPE
IS3411 ELERENRELRRSNDILRQASAYFAKAEFDRLWKKMMPLLDKLREQYGVGPLCSELHIAPSTYYHCQQQRH
220 230 240 250 260 270 280
---------.---------.---------.---------.---------.---------.---------.
IS407 PDHENEVLAARLVEL AHERRRFGYRRLHALVEREGTHANHKRIYRLYREAGLAVRRRRKR
'IS3Ea9' RPAADAHLSGRITEL ALERRRFGYRRIWQLLRREGLHVNHKRVYRLYHLSGLGVKRRRRR
IS476 PTPATQALSARLVEL AQTHRRFGYRRLHDLLRPEFPSVNHKKIYRLYEEAELKVRKRRKA
ISR1 RPDDASARPA EGP GQERRRFGYRRLHVLLRREGHAVNRKRVQRIYRDEQLTVRRRAPQ
IS150 KPDKYADVKKRISEI YHENRGRYGYRRVTLSLHREGKQINHKAVQRLMGTLSLKAAIKVKR Y
IS861 KPDKDKAIKSDIQSI YDEHRGNYGYRRIYLELRNRGFVINHKRVQGLMKSMGLTARIRRKRKY
IS3 RISTRQQFRQHCDSV VLAAFTRSKQRYGAPRLTDELRAQGYPFNVKTVAASLRRQGLRA KASRKF
IS911 KPDGRRAV LRSQVL ELHGISHGSA GARSIATMATRRGYQMGRWLAGRLMKELGLVSCQQPTHRY
IS3411 HPDKRSARAQRDDWLKKQIQRVYDENHKVYGVRKVWRQLLREGIRVARCTVARLMAVMGLAGVLRGKKVR
290 300 310 320 330 340 350
---------.---------.---------.---------.---------.---------.---------.
IS407 QGVMIE REQLALPGAPNEVWSIDFVMDALSNGRRVKCLTVVDDFTKEAVDIVVDHGISGL
'IS3Ea9' KGLATE RLPLLRPAAPNLTWSDDFVMDALATGRRIKCLTCVDDFTKECLTVTVAFGISGV
IS476 KRPVGE RQKLLASSMPNDTWSMDFVFDALANARRIKCLTVVDDFTRESVDIAVDHGISGA
ISR1 AS DG HAATVTCRWRQPALVADFVADQLTDGRRFRILTVIDNCTRECLALVADTSLSGA
IS150 R SYRGEVGQTAPNVLQRDFKATRPNEKWVTDVTEFAVNGRKLYLSP VIDLFNNEVISYSLSERPVMN
IS861 A SYKGEVGKKADNLIQRQFEGSKPYEKCYTDVTEFALPEGKLYLSP VLDGYNSEIIDFTLSRSPALK
IS3 SPVSYRAHGLPVSENLLEQDFYASGPNQKWAGDITYLRTDEGWLYLAV VIDLWSRAVIGWSMSPRMTAQ
IS911 KRGGHEHVAIP NYLERQFAVTEPNQVWCGDVTYIWTGKRWAYLAV VLDLFARKPVGWAMSFSPDSR
IS3411 TTISRKAVAAG HRVNRQFVAERPDQLWVADFTYVSTWRGFVYVAF IIDVFAGYIVGWRVSSSMETT
360 370 380 390 400 410 420
---------.---------.---------.---------.---------.---------.---------.
IS407 YVARALDRA ARFRGYPKAVRTDQGPEFTSRALDQWAYANGVTLKLIQAGKPTQNAYIESFNGKFRDE
'IS3Ea9' QVTRILDSI ALFRGYPATIRTDQGPEFTCRALDQWAFEHGVELRLIQPGKPTQNGFIESFNGRFRDE
IS476 YVVRLLDQA ACFRGYPRAVRTDNGPEFTSRAFIAWTQQHGIEHILIEPGAPTQNAYIESFNGKFRDE
ISR1 RVVRELDAV IRQRGRPDTIVSDNGTEYTSNAVLAWATTPAFGWHYIAPGKPQQNGFNESFNGRLRDE
IS150 MVENMLDQAFK KLNPHEHPVLHSDQGWQYRMRRYQNILKEHGIKQSMSAKGNCLDNAVVECFFGTLKSE
IS861 QVQTMLERAFP A ASYSETILHSDQGWQYQHKSYHQFLEDKGIRPSMSRKGNSPDNGMMESFFGILKSE
IS3 LACDALQMALW RRKRPRNVIVHTDRGGQYCSADYQAQLKRHNLRGSMSAKGCCYDNACVESFFHSLKVE
IS911 LTMKALEMA WETRGKPGGVMFHSDQGSHYTSRQFRQLLWRYQIRQSMSRRGNCWDNSPMERFFRSLKNE
IS3411 FVLDALEQALW TRSTAGTVHHSDKGSQYVSLAYTQRLKEAGLLASTGSTGDSYDNAMAESINGLYKAE
430 440 450 460 470 480 490
---------.---------.---------.---------.---------.---------.---------.
IS407 CLNEHW F TTLAHARAVIAAW RQDYNEQRPHSALNYLAPSEFAAKHR
'IS3Ea9' CLNEHW F SDVSHARKTISEW RQDYNEYRPHSALNYQAPSEFAAAWR
IS476 CLNEHW F TSLAQARDVIADW RRHYNQIRPHSSCGRIPPAQFAANYRT
ISR1 LLNETL FRSLPPLAPCGGVATRLQRAASALEARVADAAGLCRSAHRTQRPACCVVDGCADRPLANPAD
IS150 CFY LD EF SNISELKDAVTEYI EY YNSRRISLKLKGLTPIEY
IS861 MFYGLEKSY KSLDDLEQAITDYI FY YNNKRIKAKLKGLSPVQY
IS3 CIHGEH F ISREIMRATVFNYI ECDYNRWRRHSWCGGLSPEQF
IS911 WIPVVG Y VSFSEAAHAITDYI VGYYSALRPHEYNGGLPPNES
IS3411 VIHRKS W KNRAEVELATLTWV DW YNNRRLLERLGHTPPAEA
500 555
---------.-----
IS407 ATADAPAAFQELV
'IS3Ea9' KGNSDSEGSDITK
IS476 QQANNAVPFNPGLYQ
ISR1 HSSDQPRTLVMAG
IS150 RNQTYMPRV
IS861 RTKSFT
IS3 ENKNLA
IS911 ENRYWKKL
IS3411 E
__________________________________________________________________________
Abbildung 25: Potentielle orfA-orfB Fusionsproteine einer Auswahl von IS-Elementen der IS3-Familie sind in einem multiplen Alignment dargestellt. Der Übergangsbereich zwischen orfA und orfB ist fett gedruckt. Der Rasterschub in der Sequenz von Phase 0 (orfA) zu -1 (orfB) wurde am erwarteten 'Frameshift'-Fenster des jeweiligen IS-Elementes eingeführt; Erläuterungen im Text (vgl. Tab.14).
Abbildung 25 zeigte trotz der unterschiedlichen Größe der jeweiligen Übergangsbereiche eine Konservierung in der AS-Sequenz. 'IS3Ea9' weist in dieser Region Homologien zu den Übergangsbereichen von IS407, IS476 und ISR1 auf. Bei IS407 ist dieser extrem große Abschnitt über weite Bereiche homolog zum ORFB der aufgeführten IS3-Elemente. Es wurde nun auch verständlich, warum das Alignment der ORFB-Proteine im Bereich des N-Terminus eine geringere Homologie zeigte (s.u. 4.4.3), denn für ein korrektes Alignment von ORFB sind anscheinend die zusätzlichen AS der ORFA-ORFB-Übergangsbereiche notwendig.
Zwar ließen sich für diesen Übergangsbereich keine Homologien zu typischen 'Linker'-Sequenzen (meint: 'Linker' als Koppler funktioneller Untereinheiten in multimeren Proteinen) finden, doch erscheint das Ergebnis des obigen Alignments als ein schlüssiger Beleg für ein ribosomales 'Frameshifting' und die Bildung von Fusionsproteinen.
4.4.6 Sequenzhomologie zu Virulenzplasmid pIB1 von Yersinia pseudotuberculosis
Bei Datenbankrecherchen mit der AS-Sequenz von ORFB, die zum einen die hohe Homolgie zu IS-Elementen der IS3-Familie ergaben, zeigte sich ebenfalls ein hohes Maß an Übereinstimmung mit einem sequenzierten Bereich von Plasmid pIB1, das in Yersinia pseudotuberculosis für dessen Virulenzeigenschaften verantwortlich gemacht wurde [Krause et al., 1990 u. 1991].
Ein Alignment der DNA-Sequenz von 'IS3Ea9' mit der Sequenz des 1.6 kb BamHI-PstI-Fragments von pIB1 ['Accession-Number' in der EMBL/Genbank Datenbank: M58506] zeigte 61 % Identität in einem Bereich von 300 bp. Die weitere Auswertung in diesem homologen Bereich der pIB1-Sequenz ergab ein offenes Leseraster von Position 1332 bis 1631 mit 101 aa, das von auswärts in die Sequenz hereinläuft und im folgenden als orfYpb bezeichnet wurde. Im Bereich von Position 1297-1320 konnte eine hohe Ähnlichkeit zum rechten 'Inverted Repeat' von IS3-Elementen gefunden werden. Ein Homologievergleich dieser Sequenz mit der IR-Consensus-Sequenz und von ORFB der IS3-Elemente mit ORFYpb, dem potentiellen orfYpb-Translationprodukt, ist in Tabelle 15 abgebildet.
Tabelle 15: Homologievergleich von 'Inverted Repeats' und ORFB von IS3-Elementen mit pIB1
_________________________________________________________________________
A
Basenfolge
pIB1 1.6 kb BamHI-PstI-Fragment 1297- TGATCCCTCCCCAATATCCGTACC -1320
||| | |||| || |
Consensus-Sequenz TGA-CTG-CCCC--------TAGC
B
|
IS-Element |
IS407 |
IS476 |
ISR1 |
'IS3Ea9' |
ORFYpb |
|
IS407 |
100/277 |
||||
|
IS476 |
63.7/179 |
100/277 |
|||
|
ISR1 |
45.7/116 |
41.4/220 |
100/305 |
||
|
'IS3Ea9' |
66.6/176 |
54/265 |
43.6/220 |
100/276 |
|
|
ORFYpb |
54.5/99 |
45.3/106 |
47.4/57 |
51.5/97 |
100/100 |
_________________________________________________________________________
Tabelle 15: A) Oben ist die postulierte Sequenz des IR-R auf pIB1 aufgeführt, darunter die Consensus-Sequenz für IR der IS3-Familie (vgl. 4.4.1; Konserviertheit der Nukleotide im Consensus: 80 %); übereinstimmende Nukleotide wurden durch Linien verbunden. B) Die Homologie im ORFB innerhalb der IS3-Familie wurde für einige ausgewählte IS-Elemente bestimmt. Die tabellarischen Angaben bezeichnen den Grad der Identität auf AS-Ebene [%] bezogen auf die Größe der Überlappung im Alignment [aa]. Das Alignment und die anschließende Berechnung der Homologie erfolgte mit Hilfe des GCG-Programms FASTA. ORFYpb bezeichnet das offene Leseraster von Position 1332-1631.
Nach Tabelle 15 zeigte ORFYpb die höchste Homologie zu IS407 und 'IS3Ea9'; die mittlere Homologie zum ORFB der aufgeführten IS3-Elemente beträgt 50 %; dieser Wert entspricht auch den Verhältnissen innerhalb der IS3-Familie. Aufgrund der vorliegenden Auswertungen ist zu vermuten, daß in der angegebenen DNA-Sequenz von Plasmid pIB1 ein noch nicht beschriebenes IS-Element der IS3-Familie ansequenziert wurde. Eine Übersicht der Homologieverhältnisse ist in Abbildung 26 dargestellt.
Abbildung 28: Struktur eines möglichen IS3-Elementes auf pIB1 bei Yersinia pseudotuberculosis
_________________________________________________________________________
_________________________________________________________________________
Abbildung 26: A) zeigt die typische Struktur von IS3-Elementen: Die offenen Pfeile kennzeichnen orfA und orfB, IR wurden als graue Boxen markiert. B) veranschaulicht die durch Homologie-vergleiche postulierten Verhältnisse in der Sequenz von pIB1: Im sequenzierten Bereich sind die Strukturen mit durchgezogener Linie, im nicht sequenzierten Bereich der vermuteten Struktur als Punktlinie gezeichnet. Die Zahlenangaben beziehen sich auf Positionen in der Sequenz AC: M58506. Restriktionsenzyme wurden wie angegeben abgekürzt, die schraffierte Box symbolisiert einen zu IS630 homologen Bereich.
Nach Literaturangaben [Krause et al., 1990 u. 1991] liegt auf pIB1 in einer Länge von 200 bp (Position 1096-1300) ein zu IS630 homologer Bereich. Auf einem weiteren Virulenzplasmid, pSDL2 von Salmonella dublin, das nach DNA-DNA-Hybridisierungen in weiten Bereichen Homologien zu pIB1 aufwies, konnte an der gleichen Stelle ein vollständiges IS-Element mit Homologie zu IS630 gefunden werden. Bisher konnte für den Abbruch der Homologie auf pIB1 an Position 1300 keine Erklärung gefunden werden [Krause et al., 1990]. Da die Sequenz vom IS630-homologen IS-Element bei pIB1 um die Position 1300 abbricht, an Position 1297 jedoch, wie oben gezeigt, das IS-Element mit Ähnlichkeit zur IS3-Familie beginnt, besteht die Möglichkeit, daß dieses IS3-homologe IS-Element auf pIB1 in das IS630-homologe IS-Element inseriert hat. Mit der Arbeitsgruppe, die Bereiche des pIB1 sequenziert hat, wird Kontakt aufgenommen, da es von Interesse wäre zu erfahren, ob das zum 1.6 kb BamHI/PstI benachbarte PstI-Fragment von pIB1 sequenziert wurde, weil sich dann zeigen müßte, ob sich auf pIB1 das vollständiges IS3-Element befindet.
4.4.7 Phylogenetische Zuordnung von 'IS3Ea9' in die IS3-Familie
Aufgrund der Homologie in Bezug auf charakteristische Strukturmerkmale, signifikante Sequenzhomologien auf DNA- und AS-Ebene konnte das IS-Element 'IS3Ea9' der Verwandtschaftsgruppe der IS3-Familie zugeordnet werden. Um die phylogenetische Stellung innerhalb dieser Familie zu bestimmen, wurden für einige IS3-Elemente, darunter auch 'IS3Ea9', die jeweiligen Verwandtschaftsbeziehungen für die graphische Darstellung in Form eines Dendrogramms ermittelt.
Unter Verwendung des HUSAR-Programms TREE [Feng u. Doolittle, 1987] wurden zunächst die AS-Sequenzen der ORFAB-Fusionsproteine in einem multiplen Alignment zusammengestellt. Zu diesem Sequenzvergleich wurden anstelle von ORFA bzw. ORFB die ORFAB-Fusionsproteine verwendet (vgl. unter 4.4.5), da auf diese Weise der gesamte kodierende Bereich für die spätere Ermittlung der jeweiligen Verwandtschaftsbeziehung mit einbezogen wurde. Basierend auf diesem Sequenzalignment entwickelte das obige Computer-Programm TREE das in Abbildung 27 dargestellte Dendrogramm.
Abbildung 27: Stammbaum einiger ausgewählter IS3-Elemente
_________________________________________________________________________
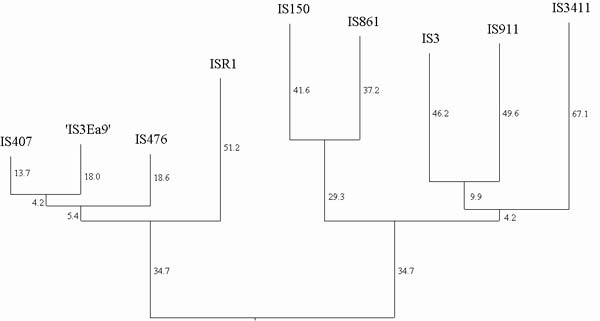
_________________________________________________________________________
Abbildung 27: Für die Erstellung des vorliegenden Dendrogramms wurde das HUSAR-Programm TREE verwendet [Feng u. Doolittle, 1987]. Ausgehend von einem Sequenzalignment der ORFAB-Fusionsproteine wurde die Ähnlichkeit der IS-Elemente in eine mathematische Zahlenmatrix umgesetzt. Die in der Graphik angegebenen Zahlenwerte stehen als Einheiten für die relative Ähnlichkeit der jeweiligen IS-Elemente.
Das Dendrogramm in Abbildung 27 zeigt, daß es sich bei der IS3-Familie um keine homogene Gruppe handelt, sondern daß sich weitere Untergruppen hoher Sequenzhomologie bilden. Beispielsweise steht 'IS3Ea9' zusammen mit den IS-Elementen IS407, IS476 und ISR1 in einer derartigen 'Unterfamilie'. Auch das unter 4.4.6 postulierte IS-Element bei Yersinia pseudotuberculosis zeigte zu IS407 die höchste Homologie und wäre dementsprechend in dieselbe 'Unterfamilie' einzuordnen.
Ein Vergleich, ob die gefundenen Verwandtschaftsbeziehungen mit den Verwandtschafts-verhältnissen der jeweiligen Wirtsorganismen korreliert ist, zeigte ein negatives Ergebnis: Obwohl die IS-Elemente IS407 und 'IS3Ea9' einen hohen Verwandtschaftsgrad zeigten, gehören die Wirtsbakterien den Gattungen Pseudomonas bzw. Enterobacter an. Andererseits kommen IS150, IS3 und IS3411 alle in E.coli vor, doch zeigte beispielsweise IS150 ein höheres Maß an Verwandtschaft mit IS861 (aus Streptococcus sp) als mit IS3 oder IS3411.
Entsprechend diesen Ergebnissen verläuft die Evolution von IS-Elementen offensichtlich unabhängig von der phylogenetischen Entwicklung jeweiliger Wirtsorganismen. Die Verbreitung bzw. Phylogenie von IS-Elementen über weite Zeiträume verläuft demnach nicht nur auf vertikaler, sondern auch in erhöhtem Maße auf horizontaler Ebene.
4.5 Vorkommen und Verbreitung von 'IS3Ea9'
4.5.1 Lokalisierung von homologen Bereichen auf pEA9
Für viele IS-Elemente ist bekannt, daß sie in einer Wirtszelle nicht einzeln, sondern oft in einer variablen, für die jeweilige Wirtsspezies charakteristischen Kopienzahl vorkommen. Beispielsweise liegt IS3 bei E.coliK12 in fünf bis sechs Kopien vor, wobei davon zwei auf dem F-Plasmid, die restlichen drei bis vier chromosomal vorliegen [Iida et al., 1983]. Um festzustellen, ob sich auch in E.aggl.339 weitere Kopien von 'IS3Ea9' befinden, wurde zunächst untersucht, ob auf dem Plasmid pEA9 Homologiebereiche zu 'IS3Ea9' existieren, bei denen es sich um mögliche Kopien handeln könnte. Die Suche nach Homologiebereichen wurde mittels DNA-DNA-Hybridisierungen mit der 'IS3Ea9'-spezifischen 0.8 SB-Sonde durchgeführt (vgl. unter 4.1), wobei mögliche Homologiebereiche nach BamHI-Restriktionsverdau auf entsprechenden Fragmenten lokalisiert werden sollten. Da es sich bei pEA9 jedoch um ein Plasmid der Größe ~200 kb handelt, war für BamHI mit einer Vielzahl von Fragmenten zu rechnen (30 BamHI-Fragmente wurden bereits beschrieben [Steibl, 1989]) was eine spätere Zuordnung von Hybridisierungssignalen erschwert hätte.
Aus diesem Grunde wurden aus der Cosmid-Genbank von pEA9 [Steibl, 1989], in der ein Großteil des Plasmids kloniert vorliegt, pHD-Klone ausgewählt. So konnten spätere Hybridisierungssignale von pEA9 den jeweiligen pHD-Klonen zugeordnet werden, deren BamHI-Inserts größtenteil kartiert sind. Um zu entscheiden, welche pHD-Klone (es existieren 17 unterschiedliche Cosmidklone von pEA9) am geeignetesten erscheinen, wurde Plasmid-DNA von pHD39, pHD41, pHD62 und pHD54, die den gesamten klonierten Bereich abdeckten (vgl. Abb.28), sowie pJJ5.4HB als Positiv- und pUC18 als Negativkontrolle mittels Dot-Blot und anschließender Hybridisierung mit der 0.8 SB-Sonde 'vorgescreent' (Ergebnis nicht abgebildet). Der Hybridisierungsfilter zeigte eindeutige Signale bei pJJ5.4HB, pHD41 und pHD54, während pUC18, pHD39 und pHD62 kein Signal gaben. Offensichtlich existierten in der klonierten Region von pEA9 Homologiebereiche, die sich auf die in pHD41 und pHD54 klonierten Bereiche eingrenzen ließen.
Zur genauen Bestimmung der Anzahl und Lage von Homologiebereichen zu 'IS3Ea9' auf pEA9 wurde zuvor gewonnene Plasmid-DNA von pEA9 und den pHD-Klonen pHD21, pHD54, pHD41, pHD10 und pHD62, die den zuvor eingegrenzten Bereich der klonierten Region in verschiedenen Überlappungsmustern ihrer Inserts abdeckten (Abb.28), mit BamHI geschnitten und gelelektrophoretisch aufgetrennt. Nach Southern Blot des Agarose-Gels wurde der Filter mit der 0.8 SB-Sonde unter hoher Stringenz hybridisiert. Das Ergebnis ist in Abbildung 29 dargestellt.
Abbildung 28: Übersicht der klonierten Region von pEA9
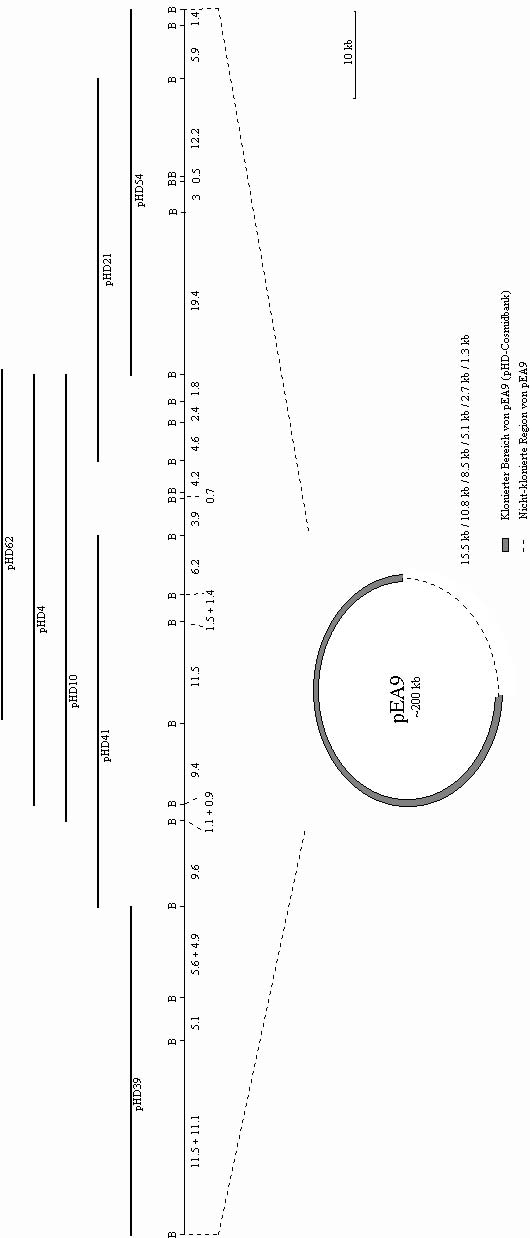
____________________________________________________________________________
Abbildung 28: pEA9 Cosmidklone [Steibl, 1989; Steibl et al., 1993]. Oberhalb der BamHI-Restriktionskarte wurden die Inserts einiger pHD-Cosmidklone, die jeweils unterhalb liegende BamHI-Fragmente tragen, als Balken dargestellt. 10 kb wurden als Maßstab angegeben. Die Zahlenangaben unter der Restriktionskarte geben die Größe der jeweiligen BamHI-Fragmente an.
Abbildung 29: Restriktionsverdau von pEA9 und pHD-Cosmidklonen mit Hybridisierungsergebnis
_________________________________________________________________________
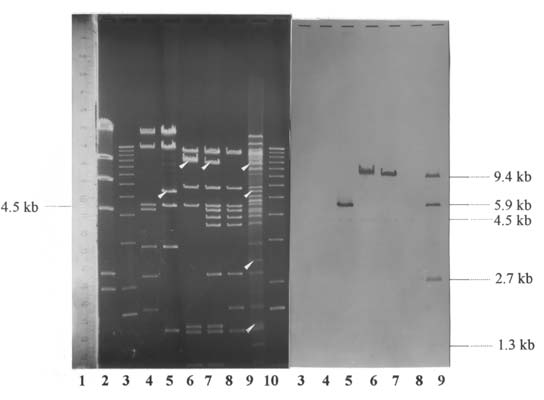
1 - Maßstab [cm] Spur 6: pHD41 + BamHI
Spur 2: l -DNA + HindIII Spur 7: pHD10 + BamHI
Spur 3: KBL Spur 8: pHD62 + BamHI
Spur 4: pHD21 + BamHI Spur 9: pEA9 + BamHI
Spur 5: pHD54 + BamHI Spur 10: KBL
_________________________________________________________________________
Abbildung 29: Links das Restriktionsmuster von pEA9 und pHD-Cosmidklonen mit cm-Maßstab, l -HindIII-Längenstandard und KBL, rechts der entsprechende Filter nach Hybridisierung mit 0.8 SB-Sonde. Die Hybridisierungssignale sind mit den jeweiligen Fragmentgrößen beschriftet, die Gelbanden mit Hybridisierungssignal durch Pfeile gekennzeichnet. Nummern unter dem Gel bzw. Filter geben die jeweilige Spur an.
Das Hybridisierungsergebnis zeigte für pEA9 Signale bei vier BamHI-Fragmenten der Größen 1.3 kb, 2.7 kb, 5.9 kb und 9-9.8 kb, von denen zwei den entsprechenden Signalen im Bereich der pHD-Klone zugeordnet werden konnten: Das 5.9 kb BamHI-Fragment von pHD54 gab das erwartete Signal. Das zweite Signal ließ sich einer großen BamHI-Doppelbande > 9 kb zuordnen, die Signalen in pHD41 und pHD10 entsprach. Somit konnte es sich hier nur um die 9.4- und 9.6 kb BamHI-Fragmente handeln, die sich im Gel nicht auftrennen ließen. Da pHD10 ein Signal gab, stand fest, daß das 9.4 kb BamHI-Fragment mit Sicherheit hybridisiert (vgl. pEA9 Übersichtskarte in Abbildung 28). Jedoch war nicht ausschließen, daß auch das 9.6 kb BamHI-Fragment in der Doppelbande bei pEA9 und pHD41 hybridisiert: Ein Cosmid-Klon, der allein das 9.6 kb Fragment trägt, existiert nicht.
Die verbliebenen zwei Signale auf pEA9 bei 1.3 und 2.7 kb fanden im Bereich der pHD-Klone keine Entsprechung; diese Homologiebereiche liegen demnach außerhalb des klonierten Bereiches von pEA9 [Steibl, 1989].
Um den Homologiebereich auf dem 9.4 kb BamHI-Fragment genauer einzugrenzen, wurde eine Restriktionskartierung von pHD4 durchgeführt; es wurde dieser Cosmidklon ausgewählt, da er das besagte Fragment am Rand des 47.6 kb-Inserts trägt, an der Grenze zum pJA1-Vektor, und eine Kartierung könnte unter Umständen Hinweise auf die Orientierung des Inserts im Vektor liefern, was eine spätere Ansequenzierung vereinfachen würde. Zur Kartierung wurden die Restriktionsenzyme EcoRI, SphI, HindIII und BamHI im Einfach- und Doppelverau verwendet; BamHI deshalb, weil damit der Anschluß an die bereits bestehende BamHI-Restriktionskarte für das Insert gewährleistet war, und EcoRI hat im pJA1-Vektormolekül, ebenso wie SphI, nur eine singuläre Schnittstelle, was die Bestimmung der Orientierung des Inserts im pHD4 ermöglichen könnte.
Die Restriktionsverdaus wurden im Agarose-Gel elektrophoretisch aufgetrennt und nach Southern Blot und Hybridisierung des Kartierungsgels mit der 0.8 SB-Sonde zeigten die folgenden Restriktionsfragmente Hybridisierungssignale: 9.4 kb BamHI, 4.0 kb BamHI/SphI, 7.0 kb SphI, 5.2 kb SphI/EcoRI, 5.2 kb EcoRI, 3.9 kb EcoRI/BamHI, 2.6 kb EcoRI/HindIII, 2.6 kb HindIII, 2.6 kb SphI/HindIII und 2.3 kb BamHI/HindIII. Das Ergebnis der Restriktionsverdaus und Hybridisierung wurde in Abbildung 30 dargestellt.
Durch Auswertung dieses Restriktions- und Hybridisierungsmusters ließ sich eine Restriktionskarte im Bereich des 9.4 kb BamHI-Fragmentes von pHD4 erstellen. Der 'IS3Ea9'-homologe Bereich konnte dabei auf das 2.3 kb BamHI-HindIII-Fragment eingegrenzt werden - an der 'Grenze' des Inserts im pJA1-Vektor: Die Größe des 5.2 kb EcoRI- und 7.0 kb SphI-Fragmentes deuteten darauf hin, daß jeweils eine HindIII- und SphI-Schnittstelle bereits im Vektor kartierten. Denn hätte der IS-homologe Bereich auf der gegenüberliegenden Seite des 9.4 kb Fragmentes gelegen, müßte das im Insert angrenzende 11.5 kb BamHI-Fragment die Restriktionsschnittstellen für SphI und HindIII im gleichen Abstand zur BamHI-Schnittstelle aufweisen wie der pJA1-Vektor. Im Gelbild müßten diese gleich großen Restriktionsfragmente als Doppelbanden sichtbar sein. Da dies nicht der Fall war, stand die Orientierung des Inserts im pHD4 fest. Die resultierende Plasmidkarte von pHD4 wurde in Abbildung 31 dargestellt.
Abbildung 30: Restriktionsverdau von pHD4 mit Hybridisierungsergebnis
_________________________________________________________________________
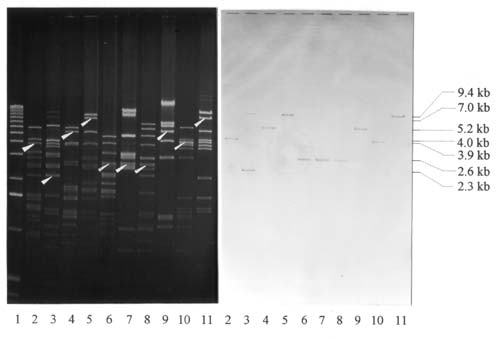
Spur 1: KBL Spur 7: pHD4 + HindIII
Spur 2: pHD4 + BamHI + SphI Spur 8: pHD4 + HindIII + EcoRI
Spur 3: pHD4 + BamHI + HindIII Spur 9: pHD4 + EcoRI
Spur 4: pHD4 + SphI + EcoRI Spur 10: pHD4 + BamHI + EcoRI
Spur 5: pHD4 + SphI Spur 11: pHD4 + BamHI
Spur 6: pHD4 + SphI + HindIII
_________________________________________________________________________
Abbildung 30: Links das Restriktionsmuster von pHD4 mit KBL, rechts der entsprechende Filter nach Hybridisierung mit 0.8 SB-Sonde. Die Hybridisierungssignale sind mit den jeweiligen Fragmentgrößen beschriftet, die Gelbanden mit Hybridisierungssignal durch Pfeile gekennzeichnet. Nummern unter dem Gel bzw. Filter geben die jeweilige Spur an.
Abbildung 31: Detailausschnitt der Plasmidkarte von pHD4
_________________________________________________________________________
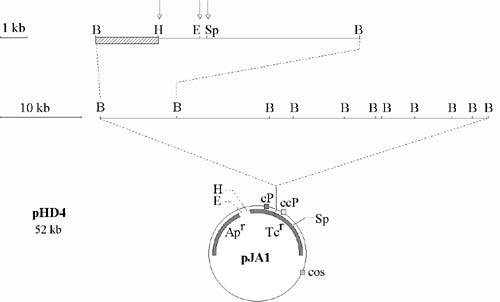
_________________________________________________________________________
Abbildung 31: Plasmidkarte von pHD4 mit pJA1 als ringförmiger Vektor und das BamHI-Insert im Maßstab 10 kb als Winkel dargestellt. Das 9.4 kb BamHI-Fragment wurde als Ausschnitt im Maßstab 1 kb vergrößert (oben), die neu kartierten Restriktionsschnittstellen mit einem Pfeil markiert. Restriktionsenzyme wurden wie folgt abgekürzt: B-BamHI, E-EcoRI, H-HindIII und Sp-SphI; cP kennzeichnet den 'clockwise'-, ccP den 'counter-clockwise'-BamHI-Primer. Der homologe Bereich zu 'IS3Ea9' konnte auf das als schraffierte Box gekennzeichnete Restriktionsfragment eingegrenzt werden.
Es ist nun möglich durch Sequenzierung des pHD4 unter Verwendung des pBR322-BamHI-'clockwise'-Primers zumindest Teile des IS-homologen Bereiches anzusequenzieren. Das Ziel wäre der konkrete Nachweis einer Kopie von 'IS3Ea9' oder einer entsprechenden Isoform durch Homologievergleiche.
4.5.2 Vorkommen von homologen Bereichen in der Gattung Enterobacter
Parallel zur Isolierung von E.aggl.339 wurden seinerzeit weitere Enterobacter-Stämme als Wildtyp-Isolate aus Rhizosphären von Weizenpflanzen aus der näheren Umgebung von Bayreuth charakterisiert [Kleeberger et al., 1983] und in weiteren Arbeitsgruppen des Lehrstuhls für Genetik untersucht. Beispielsweise gelang durch die Kurierung von E.aggl.335 und -339 (d.h., die Herstellung der kurierten Stämme E.aggl. 335/18-1 und -339/22-1) der Nachweis, daß die Fähigkeit zur Stickstoff-Fixierung plasmidkodiert auf pEA5 bzw. pEA9 vorliegt [Singh et al., 1983]. Von dem 'Laborstamm' E.aggl.339-, ursprünglich beschrieben als E.aggl.339/22-1 (s.o.), wurden die weiteren Stämme E.aggl.339-a, -b und -c hergestellt, die sich in ihren jeweiligen Antibiotikaresistenzen unterscheiden (vgl. Tab.1).
Da es sich nun bei pEA9 um ein selbsttransmissibles Plasmid handelt [Klingmüller et al., 1989] und auch IS-Elemente zumindest intraspezifisch eine hohe Mobilität via horizontalem Gentransfer aufweisen [Sawyer et al., 1987], war es naheliegend, bei E.aggl.339-verwandten Enterobacter-Stämmen weitere Kopien bzw. Isoformen von 'IS3Ea9' zu vermuten.
Um nun homologe Bereiche bei weiteren Enterobacter-Stämmen zu suchen, wurde Gesamt-DNA von E.aggl. 243, -333, -334, -335, -335/18-1, -339, -339/22-1 und Plasmid-DNA von pJJ5.4HB als Positivkontrolle in einem 'Vorscreening' nach einem Dot Blot mit der 0.8 SB-Sonde unter hoher Stringenz hybridisiert. Das Hybridisierungsergebnis ist in Abbildung 32 dargestellt.
Abbildung 32: Dot-Blot Hybridisierung von Enterobacter -Gesamt-DNA und pJJ5.4HB -Plasmid-DNA
_________________________________________________________________________
|
|
1: pJJ5.4HB 2: E.aggl.243 3: E.aggl.333 4: E.aggl.334 5: E.aggl.335 6: E.aggl.335/18-1 7: E.aggl.339 8: E.aggl.339/22-1
|
_________________________________________________________________________
Abbildung 32: Dot-Blot von E.aggl.-Gesamt-DNA nach Hybridisierung mit 0.8 SB-Sonde. Erläuterungen im Text.
Das Hybridisierungsergebnis des Dot-Blots zeigte für E.aggl.333 und -334 kein Signal, während alle anderen Proben mehr oder weniger starke Hybridisierungssignale aufwiesen. Zur weiteren Beschreibung dieser im Dot-Blot gefundenen Homologiebereiche wurde Gesamt-DNA von E.coliDH5a, E.aggl.243, -335, -335/18-1, -339, -339/22-1, -339- a/b und c mit BamHI geschnitten und gelelektrophoretisch aufgetrennt. In diesem Zusammenhang wurde E.coliDH5a mitverwendet, um eine mögliche Verbreitung außerhalb der Enterobacteriaceae in der Gattung Escherichia zu testen. Nach Southern Blot des Agarose-Gels wurde die Nylonmembran mit der 0.8 SB-Sonde unter hoher Stringenz hybridisiert. Das Hybridisierungsergebnis ist in Abbildung 33 dargestellt.
Die Auswertung von diesem Hybridisierungsergebnis zeigte bei E.coliDH5a kein Signal. Damit deutet sich in einem Vorversuch die Möglichkeit an, daß der Verbreitungsbereich von 'IS3Ea9' die Gattung Escherichia nicht mit einschließt. Zum anderen besteht die Möglichkeit, daß die 'IS3Ea9'-spezifische 0.8 SB-Sonde nicht mit anderen IS3-Elementen kreuzhybridisiert.
Bei E.aggl.243 konnte ein einzelnes Hybridisierungssignal festgestellt werden, doch da dieses Signal einem BamHI-Fragment > 20 kb entsprach, ließ sich keine Aussage auf eine Bestimmung der Anzahl von Homologiebereichen treffen; schließlich könnte ein Fragment dieser Größe mehrere Homologiebereiche, d.h., Kopien von 'IS3Ea9', beinhalten.
Die Stämme E.aggl.335 und -339, bzw. -335/18-1 und -339/22-1 (mit -339--a, -b und -c), zeigten jeweils ein identisches Signalmuster. E.aggl.335/18-1 gab ein Signal, während E.aggl.335 vier zusätzliche Signale aufwies. Dasselbe Bild ergab sich für E.aggl.339 und -339/22-1, sowie die Formen a,b und c. Da es sich bei E.aggl.335/18-1 und -339/22-1 um jeweils kurierte, d.h. plasmidfreie Stämme handelte und ihnen die vier offensichtlich plasmidalen Signale fehlten, konnte das einzelne Signal nur ein chromosomaler Homologiebereich sein.
Die vier plasmidalen Signale von E.aggl.339 wurden bereits auf pEA9 lokalisiert; es handelt sich um das 1.3 kb, 2.7 kb, 5.9 kb und 9.4 kb BamHI-Fragment von pEA9. Das fünfte (chromosomale) Signal ließ sich einem BamHI-Fragment von ca. 2 kb zuordnen.
Im Laufe der letzten Jahre kam es durch neue Ergebnisse zur Taxonomie und Klassifizierung innerhalb der Enterobacteriaceae zu einer Reihe von Umstellungen in der Nomenklatur. Mittels phänotypischer Untersuchungen und DNA-DNA-Hybridisierungen wurde die 'Enterobacter agglomerans - Erwinia Gruppe' durch Schaffung der neuen Gattung Pantoea, der die Spezies Pantoea agglomerans und Pantoea dispersa angehören, aufgelöst. Der Enterobacter agglomerans Typ-Stamm ATCC 27155 und Erwinia herbicola Typ-Stamm NCPPB 2971 wurden nun Pantoea agglomerans zugeordnet; Erwinia herbicola ATCC 14589 wurde als neuer Pantoea dispersa-Typ-Stamm klassifiziert [Gavini et al., 1983 u. 1989; Beji et al., 1988].
Abbildung 33: Restriktionsverdau von Enterobacter- und Escherichia- Gesamt-DNA mit Hybridisierungsergebnis
_________________________________________________________________________
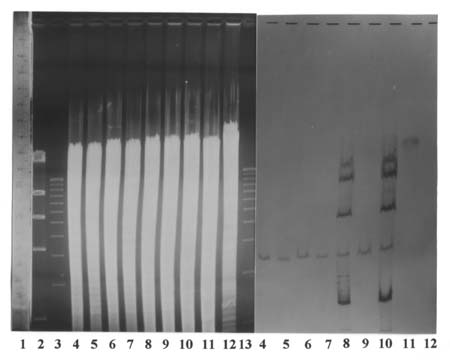
Spur 1: cm-Maßstab Spur 8: E.aggl.339 + BamHI
Spur 2: l -DNA + HindIII Spur 9: E.aggl.335/18-1 + BamHI
Spur 3: KBL Spur 10: E.aggl.335 + BamHI
Spur 4: E.aggl.339-c + BamHI Spur 11: E.aggl.243 + BamHI
Spur 5: E.aggl.339-b + BamHI Spur 12: E.coliDH5a + BamHI
Spur 6: E.aggl.339-a + BamHI Spur 13: KBL
Spur 7: E.aggl.339/22-1 + BamHI
_________________________________________________________________________
Abbildung 33: Links das Restriktionsmuster von Enterobacter- u. Escherichia-Gesamt-DNA mit cm-Maßstab, l -HindIII-Längenstandard u. KBL, rechts der entsprechende Filter nach Hybridisierung mit 0.8 SB-Sonde. Nummern unter dem Gel bzw. Filter geben die jeweilige Spur an.
Durch Verwendung physiologischer Standard-Testsysteme, basierend auf dem Prinzip des 'Enterotubes', und Bestimmung sogenannter API-Codes (Appareils et Procédés d'Identification, bioMérieux), wurden weitere Umgruppierungen von Enterobacter-Stämmen vorgeschlagen: E.aggl.339 und -335 [Kleeberger et al., 1983] sollten Rahnella aquatilis, zugeordnet werden; bei E.aggl.333 und -334 könnte es sich um Enterobacter amnigenus, E. intermedium oder ebenfalls um Rahnella aquatilis handeln [Berge et al., 1991; Korrespondenz Berge O. und Klingmüller W., 1991].
Aufgrund dieser neuer Ergebnisse zur Taxonomie der von Kleeberger beschriebenen Enterobacter agglomerans-Stämme [Kleeberger et al., 1983] war es interessant zu erfahren, ob die für eine neue Nomenklatur vorgeschlagenen Referenzstämme homologe Bereiche zu 'IS3Ea9' aufweisen.
Um mögliche Homologiebereiche zu 'IS3Ea9' in den vorgeschlagenen Referenzstämmen nachzuweisen, wurde Gesamt-DNA von E.aggl.19-78, Pantoea agglomerans und der Rahnella aquatilis-Stämme R.a.ATCC 33989, R.a.2-87, R.a.3-88 und R.a.ATCC 33071 in einem 'Vorscreening' mit E.aggl.339 als Positiv- und E.aggl.333 als Negativkontrolle nach Dot-Blot im Hybrislot-Verfahren mit der 0.8 SB-Sonde unter hoher Stringenz hybridisiert. Das Hybridisierungsergebnis ist in Abb.34 dargestellt.
Mit Ausnahme von Rahnella aquatilis ATCC 33989 und der Negativkontrolle E.aggl.333 zeigten alle Stämme ein mehr oder weniger starkes Hybridisierungssignal, was vermuten läßt, daß letztere zumindest einen Homologiebereich, d.h., eine Kopie von 'IS3Ea9', enthalten. Damit bestätigte sich auch in diesem Vorversuch die von Berge beschriebene, große Übereinstimmung zwischen den von Kleeberger charakterisierten Enterobacter- und Rahnella-Stämmen, bzw. Pantoea agglomerans [Berge et al., 1991; Kleeberger et al., 1983].
Während alle vorhergehenden Untersuchungen physiologische Übereinstimmungen aufzeigten [Berge et al., 1991], konnten hier jedoch erstmals Homologiebereiche auf DNA-Ebene gezeigt werden.
Abbildung 34: Dot-Blot Hybridisierung von Enterobacter-, Pantoea- und Rahnella-Gesamt-DNA
_________________________________________________________________________

1: E.aggl.339 5: Rahnella aquatilis 3-88
2: E.aggl.19-78 6: Rahnella aquatilis ATCC 33071
3: Rahnella aquatilis ATCC 33989 7: Pantoea agglomerans
4: Rahnella aquatilis 2-87 8: E.aggl.333
_________________________________________________________________________
Abbildung 34: Dot-Blot (im Hybrislot-Verfahren) von Enterobacter-, Pantoea- u. Rahnella-Gesamt-DNA nach Hybridisierung mit 0.8 SB-Sonde. Erläuterungen im Text, Referenzen in Tab.1.
<< nach oben
zurück
