Einleitung
Vorbemerkung
Im historischen Rückblick wurden die ersten prokaryontischen Insertionssequenzen (IS) als Ursache für Spontanmutationen bei E.coli beschrieben, während zu diesem Zeitpunkt über die Struktur mobiler DNA-Elemente und Transpositionsmechanismen als solche noch nichts bekannt war [Starlinger u. Saedler, 1976].
Nach heutiger Definition handelt es sich bei IS-Elementen um diskrete, genetische Einheiten von 0.7 bis 1.8 kb Größe mit der Fähigkeit zur erneuten Insertion in dasselbe oder ein anderes Replikon.
Diese Translokation des IS-Elementes, bezeichnet als Transposition, verläuft unabhängig von der allgemeinen, homologen Rekombination und beinhaltet (1) die Replikation des IS-Elementes und (2) das genaue Ausschneiden der DNA an den Enden des IS-Elementes und die Ligation in die Ziel-DNA, die zuvor auf spezifische Weise geöffnet wurde. Die enzymatischen Reaktionen werden dabei von der IS-eigenen Transposase katalysiert.
Die Termini aller bisher bekannten IS-Elemente zeigen als ein strukturelles Merkmal mehr oder weniger perfekte umgedrehte Sequenzwiederholungen von 10 bis 40 bp Länge, die als 'Inverted Repeats', oft abgekürzt als IR, bezeichnet werden. Es wird angenommen, daß IR bei der Fusion der IS-Enden mit der Ziel-DNA als Erkennungssequenzen für die Transposase fungieren [Iida et al., 1983]. Im Verlauf einer Insertion des IS-Elementes kommt es in der Regel an beiden Enden des IS-Elementes zu Duplikationen der Zielsequenz von 3 bis 18 bp, die als 'Direct Repeats' bezeichnet werden.
Im Rahmen einer Klassifizierung der großen Gruppe mobiler DNA-Elemente finden die IS-Elemente die weiteste Verbreitung.
Die Einteilung mobiler DNA-Elemente erfolgte in drei Klassen [Günther, 1991]:
Klasse I umfaßt sämtliche IS-Elemente und daraus zusammengesetzte Transposons, wobei das Transposase-Gen im IS-Element liegt.
Zu Klasse II gehören Transposons vom Typ des Tn3 oder Tn1000, die eigene 'Inverted Repeats' und ein eigenes Transposase-Gen haben.
Klasse III beinhaltet transponierbare Bakteriophagen, wie beispielsweise den Phagen Mu, die über jeweils komplexe und nicht immer einheitliche Strukturmerkmale verfügen.
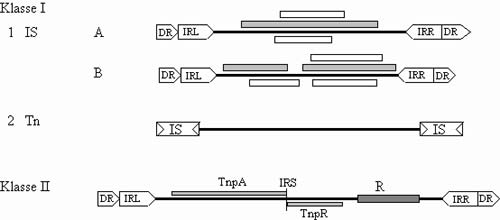
Eine schematische Übersicht zu Klasse I und -II-Strukturen ist in Abbildung 1 dargestellt.
Abbildung 1: Vergeichende Übersicht zur Struktur von IS-Elementen und Transposons
_________________________________________________________________________
_________________________________________________________________________
Abbildung 1: Übersicht mobiler DNA-Elemente [Iida et al., 1983] verändert nach Günther und Lawrence [Lawrence et al., 1991 u. 1992; Günther, 1991].
Klasse I: 1A beinhalted u.a. die IS-Elemente IS2, IS4, IS5 und IS50, während die IS-Elemente vom Typ des IS1 und IS3 Gruppe 1B angehören. Offene Leseraster, die sich in Experimenten als funktionslos herausstellten, wurden als offene Boxen und die, deren Relevanz für die Transposition nachgewiesen wurde, durch graue Boxen symbolisiert. Die Transposons der Gruppe 2 bestehen aus IS-Elementen und zusätzlichen akzessorischen Genen, wie Antibiotikaresistenzen. Klasse II: Transposons vom Typ Tn3 oder Tn1000 mit den Genen für die Cointegrase (TnpA), Resolvase/Repressor (TnpR), eigenen Resistenzmarkern (R, als dunkelgraue Box) und der internen 'Cointegrat-Resolution-Site' (IRS).
Aufgrund der Anordnung kodierender Transpositionsgene erfolgt die Einteilung von IS-Elementen in die zwei Gruppen A und B (Abb.1; [Lawrence et al., 1991 u. 1992]), wobei IS-Elemente vom Typ des IS2 (A) die Transposase in einem durchgehenden großen Leseraster von 300-400 aa kodieren. IS-Elemente der Gruppe B, IS1 und IS-Elemente der IS3-Familie, weisen zwei offene Leserahmen auf: Ein kleiner mit 80-90 aa, gefolgt von einem großen mit 260-300 aa.
Eigenschaften und Bedeutung von IS-Elementen
Durch das Fehlen eigener physiologischer Marker, wie z.B. Antibiotikaresistenzen, wurde die Erforschung von IS-Elementen zunächst sehr erschwert. Erst durch die Beobachtung, daß Transposons durch direkte oder umgedrehte Wiederholungen von IS-Elementen flankiert werden können, wie beispielsweise bei Tn9 durch direkte Wiederholung von IS1, wurden weitere Einblicke in die Biologie von IS-Elementen ermöglicht, denn die physiologisch nachweisbaren und oft selektionierbaren Marker auf den Transposons erleichterten die Untersuchung der Aktivität flankierender IS-Elemente [Calos u. Miller, 1980].
Im folgenden zeigte sich, daß IS-Elemente auf Ebene der Einzelorganismen mittels Translokation einfache bis komplexe Veränderungen im Genom mit teilweise letalen Konsequenzen induzierten. Die Insertion von IS-Elementen führte zur Unterbrechung von Strukturgenen oder starken polaren Effekten. Gelegentlich hatte die Insertion eines IS-Elementes vor ein Gen eine Aktivierung der Genexpression zur Folge; in der unmittelbaren Nachbarschaft von IS-Elementen wurde die Bildung von Deletionen stimuliert. Eine Reversion zum ursprünglichen Phänotyp wurde nach Excision dieser Elemente beobachtet [Starlinger u. Saedler, 1976].
Doch auch in der passiven Rolle als Substrat für das homologe Rekombinationssystem der Bakterienzelle sind IS-Elemente bei der Entstehung von Deletionen [Nevers u. Saedler, 1978], Inversionen [Arber et al., 1980], Cointegratbildung und anschließender Replikonfusion von Bedeutung [Galas u. Chandler, 1982]. In F- und R-Plasmiden nachgewiesene, wiederholte DNA-Sequenzen, die später als IS1, IS2, IS3 und Tn1000 beschrieben wurden, konnten durch ihre Beteiligung an Rekombinationsereignissen für massive DNA-Umstrukturierungen wie Replikonfusion bzw. -segregation, Deletionen oder Inversionen verantwortlich gemacht werden. [Davidson et al., 1975; Ohtsubo u. Ohtsubo, 1978]. So erfolgt beispielsweise die Entstehung von Hfr-Stämmen über homologe Rekombination und Cointegratbildung vom F-Plasmid mit IS2, IS3 oder Tn1000 und dem Bakterienchromosom; eine spätere Excision führt zur Entstehung von F'-Plasmiden [Davidson et al., 1975].
Für die Gesamtpopulation einer Bakterienspezies stellt dieser Mechanismus der Umstrukturierung ganzer Genombereiche, bezeichnet als Macromutation, einen Selektionsvorteil und wichtigen Evolutionsfaktor dar, denn durch eine Erhöhung der genetischen Variabilität ermöglicht er eine schnellere Anpassung an herrschende Umweltbedingungen [Syvanen, 1984]. IS-Elemente verleihen ihrem Wirt einen Selektionsvorteil, auch wenn dieser nicht immer so offensichtlich ist wie die Antibiotikaresistenz vieler Transposons. In einem Langzeitversuch im Chemostat konnte gezeigt werden, daß allein die Präsenz von IS10 in einer Bakterien-Flüssigkultur ein besseres Wachstum, relativ zur IS-freien Referenzkultur, bewirkte [Chao u. McBroom, 1985]. IS-Elemente ermöglichen über den Mechanismus der Macromutation Evolutionssprünge und Prozesse zur sympatrischen Artbildung. Durch ihre Fähigkeit zur Mobilisierung von Genen in Form von zusammengesetzten Transposons sind sie für den horizontalen Gentransfer auf inter- und intraspezifischer Ebene in großen Populationen verantwortlich.
Wie für alle biologischen 'Parasiten' ist auch das Überleben der IS-Elemente eine Gratwanderung zwischen Erhalt und Minimierung der Schäden am Wirt und der eigenen Reproduktion: Bei zu hoher Kopienzahl besteht die Gefahr, daß der Wirt durch die Veränderungen seines Genoms zugrunde geht, bei zu geringer Kopienzahl ist der Bestand des IS-Elementes gefährdet. Zu diesem Zweck haben sich bei verschiedenen IS-Elementen unterschiedliche Mechanismen zur Autoregulation und Einbeziehung von Wirtsparametern, wie dem physiologischer Zustand der Zelle, zur Steuerung der Transpositionsrate entwickelt. So kann der Schaden im Wirtsgenom durch Deletionen, Inversionen und Aktivierung von Genen in cis bzw. polare Effekte begrenzt werden [Prentki et al., 1986 u. 1987].
Bei IS10 wird die Bildung der Transposase durch eine 'Antisense'-RNA als Translationsrepressor autoreguliert, während die Dam-Methylase des Wirtes durch Methylierung der Promotorstrukturen die Transpositionsrate reduziert [Simons u. Kleckner, 1983].
Zum Schutz vor einem externen Promotor verhindern interne Terminatoren wie in IS1, IS2 und IS3 (u.a.) die IS-Gene vor Transkription. Zusätzlich werden Initiationssignale für die Translation, wie bei IS150, durch Sekundärstrukturen unzugänglich gemacht [Schwartz et al., 1988].
Weitere Wirtsfaktoren, die einen nachgewiesenen Einfluß auf die Transpositionsrate von IS-Elementen haben, sind u.a. PolA, DNA-Gyrase, Histonartige Proteine wie IHF, Rho-Faktor und texA-Mutationen, d.h., Veränderungen im RecBCD-Komplex.
Eine Zusammenfassung charakteristischer Merkmale von IS-Elementen, Transposons und jeweiligen Untergruppen findet sich in einer Reihe von Übersichtsartikeln [Calos u. Miller, 1980; Iida et al., 1983; Syvanen, 1984; Galas u. Chandler, 1989; Chandler u. Fayet, 1993 u.a.].
IS3-Familie
IS3-Elemente zeichnen sich strukturell durch ein kleines offenes Leseraster in Phase 0, gefolgt von einem großen Leseraster in Phase -1, aus, das als orfA bzw. orfB bezeichnet wird. Im Gegensatz zu IS1, das eine gleiche Struktur aufweist, haben IS3-Elemente jedoch eine Größe von 1.2 bis 1.3 kb. IS1 hingegen ist mit 768 bp eines der kleinsten IS-Elemente und zeigt auch auf Sequenzebene keine Homologie zur IS3-Familie [Polard et al., 1991]. Zur Beschreibung der Struktur haben sich die in Abbildung 2 aufgeführten Konventionen für IS3-Elemente eingebürgert.
Abbildung 2: Konventionen zu Strukturmerkmalen der IS3-Familie
_________________________________________________________________________
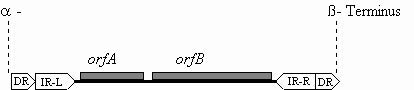
_________________________________________________________________________
Abbildung 2: In dieser schematischen Strukturübersicht von IS3-Elementen sind 'Direct Repeats' als DR, 'Inverted Repeats' als IR und offene Leseraster als graue Balken gekennzeichnet. Die Termini a u. b markieren die Enden der IS-Elemente.
Die IS-Elemente werden entsprechend der orfA- orfB-Transkriptionsrichtung von 5' (links) nach 3' (rechts) geschrieben, so daß orfA links von orfB zu liegen kommt. Die 'Inverted Repeats' werden als IR-L (links) und IR-R (rechts), die Genprodukte der Leserahmen orfA und orfB als ORFA bzw. ORFB bezeichnet [Deonier, 1987; Polard et al., 1991; Chandler u. Fayet, 1993].
Vergleichende Untersuchungen an nif-Plasmiden in Enterobacter agglomerans
1983 wurden aus Rhizosphären-Bereichen von Weizenpflanzen Bakterienstämme der Spezies Enterobacter agglomerans isoliert, welche sich u.a. durch ihre Fähigkeit zur Stickstoff-Fixierung auszeichneten [Kleeberger et al., 1983]; die dazu notwendigen nif-Gene wurden auf 100-200 kb großen Plasmiden lokalisiert [Singh et al., 1983].
Bei vergleichenden Untersuchungen der nif-Plasmide pEA3 [Kreutzer, 1986] und pEA9 von Enterobacter agglomerans 333, bzw. -339 konnten in unserer Arbeitsgruppe durch DNA-DNA-Hybridisierungsversuche Homologien im Bereich der nif-Region gefunden werden. Untersuchungen der Gesamtorganisation der jeweiligen nif-Cluster zeigten, daß die nif-Cluster beider Plasmide mit einer mittleren Sequenzhomologie von 98 % auf DNA-Ebene praktisch identisch waren. Wie Sequenzauswertungen zeigten, erfolgt der Abbruch der Homologie unmittelbar angrenzend an die nif-Region, auf pEA9 in 3'-Richtung von nifJ, was einen Verbreitungsmechanismus des nif-Clusters über horizontalen Gentransfer nahelegte [Steibl et al., 1993]. Um dies konkret abzuleiten, wurden die Randbereiche des nif-Clusters näher untersucht.
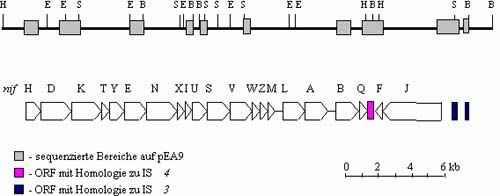
Für diese Untersuchungen stand eine BamHI-Cosmidbank des 200 kb-Plasmids pEA9 zur Verfügung [Steibl, 1989], wobei der Cosmidklon pHD54 die vollständige nif-Region trägt (Abb.3).
Abbildung 3: pEA9 Nif-Cluster
_________________________________________________________________________
_________________________________________________________________________
Abbildung 3: In der Genkarte vom pEA9-nif-Cluster symbolisieren die offenen Pfeile die jeweiligen nif-Gene. Oberhalb der Genkarte wurde die entsprechende Restriktionskarte dieser Region, kloniert in pHD54, im Maßstab 6 kb dargestellt. Sequenzierte DNA-Bereiche wurden mit grauen Boxen markiert. Zur besseren Übersicht wurden nur die kartierten Restriktionsschnittstellen für BamHI (B), EcoRI (E), HindIII (H) und SalI (S) eingezeichnet. [Darstellung nach Steibl, Mitteilung 1993].
Zur Charakterisierung der 3'-flankierenden Region des nif-Clusters von pEA9 wurden Restriktionsfragmente von pHD54 zur Sequenzierung in pUC18 kloniert. Die Ansequenzierung des 0.8 kb SalI-BamHI- und 5.4 kb HindIII-BamHI-Fragmentes und anschließende Sequenzanalysen ergaben zwei offene Leseraster, die nach Datenbank-recherchen eine Homologie von 61 % zum ORFA und 62 % zum ORFB von IS407, einem IS-Element der IS3-Familie, zeigten. Nach weiteren Analysen auf DNA-Ebene wurde in 5'-Richtung von diesem 'ORFA' ein s70 -Promotor und ein IR-L beschrieben [Steibl, Dissertation (in Vorbereitung)].
Für das postulierte IS-Element wird die vorläufige Bezeichnung 'IS3Ea9' vorgeschlagen, da sich im Vorfeld dieser Diplomarbeit eine Verwandtschaft zu IS3-Elementen zeigte und die Erstbeschreibung in Enterobacter agglomerans 339 erfolgte.
Aufbauend auf den oben genannten Ergebnissen war es das Ziel der vorliegenden Diplomarbeit, das postulierte IS-Element durch Sequenzierung genetisch zu charakterisieren und die mögliche Verbreitung in Enterobacter agglomerans 339, ggf. darüber hinaus, mittels DNA-DNA-Hybridisierungen zu untersuchen.
<< nach oben
zurück